Biologia per il liceo/La duplicazione del DNA
La scoperta del DNA
[modifica | modifica sorgente]
La nostra attuale comprensione del DNA ha avuto inizio con la scoperta degli acidi nucleici, seguita dallo sviluppo del modello a doppia elica. Negli anni '60 dell'Ottocento, Friedrich Miescher, medico di professione, isolò sostanze chimiche ricche di fosfati dai globuli bianchi (leucociti). Chiamò queste sostanze chimiche (che alla fine sarebbero state note come DNA) nucleina perché erano isolate dai nuclei delle cellule.
Mezzo secolo dopo, nel 1928, il batteriologo britannico Frederick Griffith riportò la prima dimostrazione di trasformazione batterica , un processo in cui il DNA esterno viene assorbito da una cellula, modificandone così la morfologia e la fisiologia. Griffith condusse i suoi esperimenti con lo Streptococcus pneumoniae, un batterio che causa la polmonite. Griffith lavorò con due ceppi di questo batterio chiamati rugoso (R) e liscio (S). (I due tipi di cellule furono chiamati "rugoso" e "liscio" dopo l'aspetto delle loro colonie cresciute su una piastra di agar nutriente.)
Il ceppo R è non patogeno (non causa malattie). Il ceppo S è patogeno (che causa malattie) e ha una capsula esterna alla sua parete cellulare. La capsula consente alla cellula di sfuggire alle risposte immunitarie del topo ospite.
Quando Griffith iniettò il ceppo S vivo nei topi, questi morirono di polmonite. Al contrario, quando Griffith iniettò il ceppo R vivo nei topi, questi sopravvissero. In un altro esperimento, quando iniettò nei topi il ceppo S ucciso dal calore, anche questi sopravvissero. Questo esperimento dimostrò che la capsula da sola non era la causa della morte. In una terza serie di esperimenti, una miscela di ceppo R vivo e ceppo S ucciso dal calore fu iniettata nei topi e, con sua sorpresa, i topi morirono. Dopo aver isolato i batteri vivi dal topo morto, fu recuperato solo il ceppo S dei batteri. Quando questo ceppo S isolato fu iniettato nei topi freschi, i topi morirono. Griffith concluse che qualcosa era passato dal ceppo S ucciso dal calore al ceppo R vivo e lo aveva trasformato nel ceppo S patogeno. Chiamò questo il principio di trasformazione ( Figura sotto). Questi esperimenti sono ora noti come esperimenti di trasformazione di Griffith.

Gli scienziati Oswald Avery, Colin MacLeod e Maclyn McCarty (1944) erano interessati a esplorare ulteriormente questo principio trasformativo. Isolarono il ceppo S dai topi morti e isolarono le proteine e gli acidi nucleici (RNA e DNA) poiché erano possibili candidati per la molecola dell'ereditarietà. Utilizzarono enzimi che degradavano specificamente ogni componente e poi utilizzarono ogni miscela separatamente per trasformare il ceppo R. Scoprirono che quando il DNA veniva degradato, la miscela risultante non era più in grado di trasformare i batteri, mentre tutte le altre combinazioni erano in grado di trasformare i batteri. Ciò li portò a concludere che il DNA era il principio trasformante.
-
Esperimento di Griffith
-
Oswald Avery
-
Avery comprese che erano gli acidi nucleici a rebdere patogeni i batteri
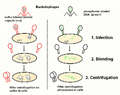
Sebbene gli esperimenti di Avery, McCarty e McLeod avessero dimostrato che il DNA era la componente informativa trasferita durante la trasformazione, il DNA era ancora considerato una molecola troppo semplice per trasportare informazioni biologiche. Le proteine, con i loro 20 diversi amminoacidi, erano considerate candidate più probabili. L'esperimento decisivo, condotto da Martha Chase e Alfred Hershey nel 1952, fornì prove confermative del fatto che il DNA era effettivamente il materiale genetico e non le proteine. Chase e Hershey stavano studiando un batteriofago , un virus che infetta i batteri. I virus hanno in genere una struttura semplice: un rivestimento proteico, chiamato capside, e un nucleo di acido nucleico che contiene il materiale genetico (DNA o RNA). Il batteriofago infetta la cellula batterica ospite attaccandosi alla sua superficie, quindi inietta i suoi acidi nucleici all'interno della cellula. Il DNA del fago crea più copie di se stesso utilizzando i macchinari dell'ospite e alla fine la cellula ospite scoppia, rilasciando un gran numero di batteriofagi. Hershey e Chase hanno selezionato elementi radioattivi che avrebbero specificamente distinto la proteina dal DNA nelle cellule infette. Hanno etichettato un lotto di fagi con zolfo radioattivo, 35 S, per etichettare il rivestimento proteico. Un altro lotto di fagi è stato etichettato con fosforo radioattivo, 32 P. Poiché il fosforo si trova nel DNA, ma non nelle proteine, il DNA e non le proteine sarebbero stati etichettati con fosforo radioattivo. Allo stesso modo, lo zolfo è assente dal DNA, ma presente in diversi amminoacidi come metionina e cisteina. A ogni lotto di fago è stato permesso di infettare le cellule separatamente. Dopo l'infezione, la sospensione batterica del fago è stata messa in un frullatore, che ha causato il distacco del rivestimento del fago dalla cellula ospite. Le cellule esposte abbastanza a lungo da causare l'infezione sono state quindi esaminate per vedere quale delle due molecole radioattive era entrata nella cellula. Il fago e la sospensione batterica sono stati centrifugati. Le cellule batteriche più pesanti si sono depositate e hanno formato un pellet, mentre le particelle di fago più leggere sono rimaste nel supernatante. Nella provetta che conteneva il fago marcato con 35 S, il supernatante conteneva il fago marcato radioattivamente, mentre nel pellet non è stata rilevata alcuna radioattività. Nella provetta che conteneva il fago marcato con 32 P, la radioattività è stata rilevata nel pellet che conteneva le cellule batteriche più pesanti e non è stata rilevata alcuna radioattività nel supernatante. Hershey e Chase conclusero che era il DNA del fago ad essere iniettato nella cellula e a trasportare informazioni per produrre più particelle fagiche, fornendo così la prova che il DNA era il materiale genetico e non le proteine ( vedi galleria ).
-
Martha Chase e Alfred Hershey
-
Il batteriofago T2 usato nell'esperimento
-
Schema dell'esperimento di Hershey e Chase
-
Schema un po' più dettagliato dell'esperimento di Hershey e Chase
-
La quantità di A = T e quella di G = C (regola di Chargaff)
Nello stesso periodo, il biochimico austriaco Erwin Chargaff esaminò il contenuto di DNA in diverse specie e scoprì che le quantità di adenina, timina, guanina e citosina non si trovavano in quantità uguali e che le concentrazioni relative delle quattro basi nucleotidiche variavano da specie a specie, ma non all'interno dei tessuti dello stesso individuo o tra individui della stessa specie. Scoprì anche qualcosa di inaspettato: che la quantità di adenina era uguale alla quantità di timina e la quantità di citosina era uguale alla quantità di guanina (ovvero, A = T e G = C). Specie diverse avevano quantità uguali di purine (A+G) e pirimidine (T + C), ma diversi rapporti tra A+T e G+C. Queste osservazioni divennero note come regole di Chargaff . Le scoperte di Chargaff si rivelarono immensamente utili quando Watson e Crick si prepararono a proporre il loro modello di doppia elica del DNA! Dopo aver letto le ultime pagine, potrete constatare come la scienza si basi sulle scoperte precedenti, a volte in un processo lento e laborioso.
La struttura del DNA
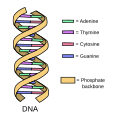
[modifica | modifica sorgente]I mattoni del DNA sono i nucleotidi. I componenti importanti del nucleotide sono una base azotata (che contiene azoto), uno zucchero a 5 atomi di carbonio (pentoso) e un gruppo fosfato. Il nucleotide è denominato in base alla base azotata. La base azotata può essere una purina come adenina (A) e guanina (G), o una pirimidina come citosina (C) e timina (T).

Esercizio: Le immagini sopra illustrano le cinque basi del DNA e dell'RNA. Esamina le immagini e spiega perché sono chiamate "basi azotate". In che modo le purine sono diverse dalle pirimidine? In che modo una purina o una pirimidina è diversa da un'altra, ad esempio, l'adenina dalla guanina? In che modo un nucleoside è diverso da un nucleotide?
Le purine hanno una struttura ad anello doppio con un anello a sei elementi fuso con un anello a cinque elementi. Le pirimidine sono più piccole; hanno una struttura ad anello singolo a sei elementi.
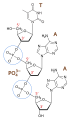
Lo zucchero è il desossiribosio nel DNA e il ribosio nell'RNA. Gli atomi di carbonio dello zucchero a cinque atomi di carbonio sono numerati 1', 2', 3', 4' e 5' (1' si legge come "un primo"). Il fosfato, che rende acidi il DNA e l'RNA, è collegato al carbonio 5' dello zucchero tramite la formazione di un legame estereo tra l'acido fosforico e il gruppo 5'-OH (un estere è un acido + un alcol). Nei nucleotidi del DNA, il carbonio 3' dello zucchero desossiribosio è legato a un gruppo idrossilico (OH). Nei nucleotidi dell'RNA, il carbonio 2' dello zucchero ribosio contiene anche un gruppo idrossilico. La base è legata al carbonio 1' dello zucchero.
I nucleotidi si combinano tra loro per produrre legami fosfodiesterici. Il residuo di fosfato attaccato al carbonio 5' dello zucchero di un nucleotide forma un secondo legame estereo con il gruppo idrossilico del carbonio 3' dello zucchero del nucleotide successivo, formando così un legame fosfodiesterico 5'-3'. In un polinucleotide, un'estremità della catena ha un fosfato 5' libero e l'altra estremità ha un 3'-OH libero. Queste sono chiamate estremità 5' e 3' della catena. Una base attaccata a uno zucchero a cinque atomi di carbonio rappresenta un nucleoside. Un nucleotide è composto da uno, due o tre gruppi fosfato attaccati a un nucleoside.
Negli anni '50, Francis Crick e James Watson lavorarono insieme per determinare la struttura del DNA presso l'Università di Cambridge, in Inghilterra. Anche altri scienziati come Linus Pauling e Maurice Wilkins stavano esplorando attivamente questo campo. Pauling aveva precedentemente scoperto la struttura secondaria delle proteine utilizzando la cristallografia a raggi X. Nel laboratorio di Wilkins, la ricercatrice Rosalind Franklin stava utilizzando metodi di diffrazione dei raggi X per comprendere la struttura del DNA. Watson e Crick furono in grado di ricomporre il puzzle della molecola del DNA sulla base dei dati di Franklin perché Crick aveva anche studiato la diffrazione dei raggi X. Nel 1962, James Watson, Francis Crick e Maurice Wilkins ricevettero il premio Nobel per la medicina. Sfortunatamente, a quel tempo Franklin era morta e i premi Nobel non vengono assegnati postumi.
Watson e Crick hanno proposto che il DNA sia costituito da due filamenti che sono attorcigliati l'uno intorno all'altro per formare un'elica destrorsa. L'appaiamento delle basi avviene tra una purina e una pirimidina su filamenti opposti, in modo che A si appaia con T e G si appaia con C (suggerito dalle regole di Chargaff). Quindi, adenina e timina sono coppie di basi complementari e anche citosina e guanina sono coppie di basi complementari. Le coppie di basi sono stabilizzate da legami idrogeno: adenina e timina formano due legami idrogeno e citosina e guanina formano tre legami idrogeno. I due filamenti sono di natura antiparallela; ovvero, l'estremità 3' di un filamento è rivolta verso l'estremità 5' dell'altro filamento. Lo zucchero e il fosfato dei nucleotidi formano la spina dorsale della struttura, mentre le basi azotate sono impilate all'interno, come i pioli di una scala. Ogni coppia di basi è separata dalla coppia di basi successiva da una distanza di 0,34 nm e ogni giro dell'elica misura 3,4 nm. Pertanto, sono presenti 10 coppie di basi per giro dell'elica. Il diametro della doppia elica del DNA è di 2 nm ed è uniforme in tutto. Solo l'appaiamento tra una purina e una pirimidina e l'orientamento antiparallelo dei due filamenti di DNA possono spiegare il diametro uniforme. La torsione dei due filamenti l'uno attorno all'altro determina la formazione di solchi maggiori e minori uniformemente distanziati.
-
Il DNA si trova nel nucleo della cellula
-
schema sintetico del DNA
-
Struttura 3d del DNA
-
Lo scheletro zucchero-fosfato
-
Legame fosfodiesterico
-
Struttura del DNA
-
Che cosa indicano i numeri?
-
Struttura del DNA
-
Dimensioni del DNA
-
modello 3d del DNA
-
Differenza tra DNA e RNA
-
Nel DNA i due filamenti sono antiparalleli
-
Rosalind Franklin
-
La famosa foto 51
-
La foto 51 e il modello 3d del DNA
-
James Watson
-
Francis Crick
-
Il modello di Watson e Crick
Avvolgimento del DNA nelle cellule
[modifica | modifica sorgente]
I procarioti sono molto più semplici degli eucarioti in molte delle loro caratteristiche ( Figura sopra). La maggior parte dei procarioti contiene un singolo cromosoma circolare che si trova in un'area del citoplasma chiamata regione nucleoide .

Un eucariote contiene un nucleo ben definito, mentre nei procarioti il cromosoma si trova nel citoplasma in un'area chiamata nucleoide.
Esercizio: Nelle cellule eucariotiche, la sintesi di DNA e RNA avviene in un compartimento separato dalla sintesi proteica. Nelle cellule procariotiche, entrambi i processi avvengono insieme. Quali vantaggi potrebbero esserci nel separare i processi? Quali vantaggi potrebbero esserci nel farli avvenire insieme?
La dimensione del genoma in uno dei procarioti più studiati, l'Escherichia coli, è di 4,6 milioni di coppie di basi (circa 1,1 mm, se tagliato e allungato). Quindi, come si adatta questo all'interno di una piccola cellula batterica? Il DNA è attorcigliato da ciò che è noto come superavvolgimento. Il superavvolgimento suggerisce che il DNA è o "sotto-avvolto" (meno di un giro dell'elica per 10 coppie di basi) o "sovra-avvolto" (più di 1 giro per 10 coppie di basi) rispetto al suo normale stato rilassato. È noto che alcune proteine sono coinvolte nel superavvolgimento; altre proteine ed enzimi come la DNA girasi aiutano a mantenere la struttura superavvolta.
Gli eucarioti, i cui cromosomi sono costituiti ciascuno da una molecola di DNA lineare, impiegano un diverso tipo di strategia di impacchettamento per adattare il loro DNA all'interno del nucleo ( Figura 14.11 ). Al livello più elementare, il DNA è avvolto attorno a proteine note come istoni per formare strutture chiamate nucleosomi. Gli istoni sono proteine evolutivamente conservate che sono ricche di amminoacidi basici e formano un ottamero composto da due molecole di ciascuno dei quattro diversi istoni. La loro composizione e proprietà sono importanti per comprendere l'espressione genica e sono state parzialmente scoperte sulla base della ricerca di Marie M. Daly e Alfred E. Mirsky nei primi anni '50. Il DNA (ricorda, è caricato negativamente a causa dei gruppi fosfato) è avvolto strettamente attorno al nucleo dell'istone. Questo nucleosoma è collegato al successivo con l'aiuto di un DNA di collegamento . Questa è anche nota come struttura "perline su una corda". Con l'aiuto di un quinto istone, una serie di nucleosomi viene ulteriormente compattata in una fibra di 30 nm, che è il diametro della struttura. I cromosomi in metafase vengono ulteriormente condensati dall'associazione con proteine di impalcatura. Nella fase di metafase, i cromosomi sono al massimo della loro compattezza, circa 700 nm di larghezza.
In interfase, i cromosomi eucariotici hanno due regioni distinte che possono essere distinte tramite colorazione. La regione strettamente impacchettata è nota come eterocromatina, e la regione meno densa è nota come eucromatina. L'eterocromatina di solito contiene geni che non sono espressi e si trova nelle regioni del centromero e dei telomeri. L'eucromatina di solito contiene geni che sono trascritti, con DNA impacchettato attorno ai nucleosomi ma non ulteriormente compattato.

La duplicazione (o replicazione) del DNA
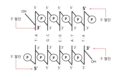
[modifica | modifica sorgente]La spiegazione della struttura della doppia elica ha fornito un indizio su come il DNA si divide e fa copie di se stesso. Nel loro articolo del 1953, Watson e Crick hanno scritto un incredibile eufemismo: "Non ci è sfuggito che l'appaiamento specifico che abbiamo postulato suggerisce immediatamente un possibile meccanismo di copia per il materiale genetico". Con coppie di basi specifiche, la sequenza di un filamento di DNA può essere prevista dal suo complemento. Il modello a doppia elica suggerisce che i due filamenti della doppia elica si separano durante la replicazione e ogni filamento funge da modello da cui viene copiato il nuovo filamento complementare. Ciò che non era chiaro era come avveniva la replicazione. Sono stati suggeriti tre modelli ( Figura sotto): conservativo, semi-conservativo e dispersivo .

Nella replicazione conservativa, il DNA parentale rimane unito e i filamenti figli appena formati sono uniti. Il metodo semi-conservativo suggerisce che ciascuno dei due filamenti di DNA parentale agisca come stampo per la sintesi di nuovo DNA; dopo la replicazione, ogni DNA a doppio filamento include un filamento parentale o "vecchio" e un filamento "nuovo". Nel modello dispersivo, entrambe le copie di DNA hanno segmenti a doppio filamento di DNA parentale e DNA appena sintetizzato intervallati.
Durante la replicazione del DNA, ciascuno dei due filamenti che compongono la doppia elica funge da stampo da cui vengono copiati nuovi filamenti. I nuovi filamenti saranno complementari ai filamenti parentali o "vecchi". Quando si formano due copie di DNA figlie, hanno la stessa sequenza e sono divise equamente nelle due cellule figlie.

La duplicazione del DNA nei procarioti
[modifica | modifica sorgente]La replicazione del DNA è stata ampiamente studiata nei procarioti principalmente a causa delle piccole dimensioni del genoma e della grande varietà di mutanti disponibili. L'Escherichia coli ha 4,6 milioni di coppie di basi in un singolo cromosoma circolare e tutto viene replicato in circa 42 minuti, partendo da un singolo sito lungo il cromosoma e procedendo attorno al cerchio in entrambe le direzioni. Ciò significa che vengono aggiunti circa 1000 nucleotidi al secondo. Quindi, il processo è piuttosto rapido e avviene senza molti errori.
La replicazione del DNA impiega un gran numero di proteine strutturali ed enzimi, ognuno dei quali svolge un ruolo critico durante il processo. Uno degli attori chiave è l'enzima DNA polimerasi , noto anche come DNA pol, che aggiunge nucleotidi uno alla volta alla catena di DNA in crescita che è complementare al filamento stampo. L'aggiunta di nucleotidi richiede energia; questa energia è ottenuta dai nucleosidi trifosfati ATP, GTP, TTP e CTP. Come l'ATP, gli altri NTP (nucleosidi trifosfati) sono molecole ad alta energia che possono servire sia come fonte di nucleotidi del DNA sia come fonte di energia per guidare la polimerizzazione. Quando il legame tra i fosfati viene "rotto", l'energia rilasciata viene utilizzata per formare il legame fosfodiesterico tra il nucleotide in arrivo e la catena in crescita. Nei procarioti, sono noti tre tipi principali di polimerasi: DNA pol I, DNA pol II e DNA pol III. Ora è noto che DNA pol III è l'enzima necessario per la sintesi del DNA; La DNA pol I è un importante enzima accessorio nella replicazione del DNA e, insieme alla DNA pol II, è necessaria principalmente per la riparazione.
-
Origine della replicazione del DNA (Aː procarioti; Bː eucarioti)
-
Duplicazione del DNA batterico (procariote) circolare
-
Schema globale della duplicazione
-
DNA polimerasi III (complesso)
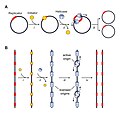
Come fa il meccanismo di replicazione a sapere da dove iniziare? Si scopre che ci sono sequenze nucleotidiche specifiche chiamate origini di replicazione dove inizia la replicazione. In E. coli, che ha una singola origine di replicazione sul suo cromosoma (come la maggior parte dei procarioti), questa origine di replicazione è lunga circa 245 coppie di basi ed è ricca di sequenze AT. L'origine di replicazione è riconosciuta da alcune proteine che si legano a questo sito. Un enzima chiamato elicasi svolge il DNA rompendo i legami idrogeno tra le coppie di basi azotate. L'idrolisi dell'ATP è richiesta per questo processo. Quando il DNA si apre, si formano strutture a forma di Y chiamate forcelle di replicazione . Due forcelle di replicazione si formano all'origine di replicazione e queste si estendono bidirezionalmente man mano che la replicazione procede. Le proteine di legame a singolo filamento rivestono i singoli filamenti di DNA vicino alla forcella di replicazione per impedire al DNA a singolo filamento di riavvolgersi in una doppia elica.
La DNA polimerasi ha due importanti restrizioni: è in grado di aggiungere nucleotidi solo nella direzione 5'-3' (un nuovo filamento di DNA può essere esteso solo in questa direzione). Richiede anche un gruppo 3'-OH libero a cui può aggiungere nucleotidi formando un legame fosfodiesterico tra l'estremità 3'-OH e il fosfato 5' del nucleotide successivo. Ciò significa essenzialmente che non può aggiungere nucleotidi se non è disponibile un gruppo 3'-OH libero. Quindi come aggiunge il primo nucleotide? Il problema viene risolto con l'aiuto di un primer che fornisce l'estremità 3'-OH libera. Un altro enzima, la RNA primasi , sintetizza un segmento di RNA lungo circa cinque-dieci nucleotidi e complementare al DNA stampo. Poiché questa sequenza innesca la sintesi del DNA, è opportunamente chiamata primer . La DNA polimerasi può ora estendere questo primer di RNA, aggiungendo uno alla volta i nucleotidi complementari al filamento stampo ( Figura 14.14 ).

La forcella di replicazione si muove alla velocità di 1000 nucleotidi al secondo. La topoisomerasi impedisce l'avvolgimento eccessivo della doppia elica del DNA davanti alla forcella di replicazione mentre il DNA si apre; lo fa causando tagli temporanei nell'elica del DNA e quindi risigillandola. Poiché la DNA polimerasi può estendersi solo nella direzione 5'-3' e poiché la doppia elica del DNA è antiparallela , c'è un piccolo problema alla forcella di replicazione. I due filamenti di DNA stampo hanno orientamenti opposti: un filamento è nella direzione 5'-3' e l'altro è orientato nella direzione 3'-5'. Solo un nuovo filamento di DNA, quello complementare al filamento di DNA parentale 3'-5', può essere sintetizzato continuamente verso la forcella di replicazione. Questo filamento sintetizzato continuamente è noto come filamento guida . L'altro filamento, complementare al DNA parentale 5'-3', si estende lontano dalla forcella di replicazione, in piccoli frammenti noti come frammenti di Okazaki , ognuno dei quali richiede un primer per avviare la sintesi. Nuovi segmenti di primer vengono depositati nella direzione della forcella di replicazione, ma ognuno punta lontano da essa. (I frammenti di Okazaki prendono il nome dallo scienziato giapponese che li ha scoperti per primo. Il filamento con i frammenti di Okazaki è noto come filamento ritardato .)
Il filamento principale può essere esteso da un singolo primer, mentre il filamento ritardato necessita di un nuovo primer per ciascuno dei frammenti corti di Okazaki. La direzione complessiva del filamento ritardato sarà da 3' a 5', e quella del filamento principale da 5' a 3'. Una proteina chiamata morsetto scorrevole mantiene la DNA polimerasi in posizione mentre continua ad aggiungere nucleotidi. Il morsetto scorrevole è una proteina a forma di anello che si lega al DNA e mantiene la polimerasi in posizione. Man mano che la sintesi procede, i primer di RNA vengono sostituiti dal DNA. I primer vengono rimossi dall'attività esonucleasica della DNA pol I, che usa il DNA dietro l'RNA come proprio primer e riempie gli spazi lasciati dalla rimozione dei nucleotidi di RNA aggiungendo nucleotidi di DNA. Le intaccature rimaste tra il DNA appena sintetizzato (che ha sostituito il primer dell'RNA) e il DNA sintetizzato in precedenza vengono sigillate dall'enzima DNA ligasi , che catalizza la formazione di legami fosfodiesterici tra l'estremità 3'-OH di un nucleotide e l'estremità 5' fosfato dell'altro frammento.
Una volta che il cromosoma è stato completamente replicato, le due copie del DNA si spostano in due cellule diverse durante la divisione cellulare.
Il processo di replicazione del DNA può essere riassunto come segue:
- Il DNA si svolge all'origine della replicazione.
- L'elicasi apre le forcelle di replicazione che formano il DNA, che vengono estese in modo bidirezionale.
- Le proteine leganti il singolo filamento rivestono il DNA attorno alla forcella di replicazione per impedirne il riavvolgimento.
- La topoisomerasi si lega nella regione antistante la forcella di replicazione per impedire il superavvolgimento.
- La primasi sintetizza i primer di RNA complementari al filamento di DNA.
- La DNA polimerasi III inizia ad aggiungere nucleotidi all'estremità 3'-OH del primer.
- Continua l'allungamento sia del filamento ritardato che di quello principale.
- I primer dell'RNA vengono rimossi dall'attività esonucleasica.
- Le lacune vengono colmate dalla DNA pol I aggiungendo dNTP.
- Lo spazio tra i due frammenti di DNA viene sigillato dalla DNA ligasi, che favorisce la formazione di legami fosfodiesterici.
La tabella sotto riassume gli enzimi coinvolti nella replicazione del DNA procariotico e le funzioni di ciascuno.
Replicazione del DNA procariotico: enzimi e loro funzione
| Enzima/proteina | Funzione specifica |
|---|---|
| DNA pol. I | Rimuove il primer dell'RNA e lo sostituisce con il DNA appena sintetizzato |
| DNA pol III | Enzima principale che aggiunge nucleotidi nella direzione 5'-3' |
| Elicasi | Apre l'elica del DNA rompendo i legami idrogeno tra le basi azotate |
| Ligasi | Sigilla gli spazi tra i frammenti di Okazaki per creare un filamento di DNA continuo |
| Primase | Sintetizza i primer di RNA necessari per avviare la replicazione |
| Morsetto scorrevole | Aiuta a mantenere la DNA polimerasi in posizione quando vengono aggiunti nucleotidi |
| Topoisomerasi | Aiuta ad alleviare la tensione sul DNA durante lo svolgimento causando rotture e quindi risigillando il DNA |
| Proteine leganti a singolo filamento (SSB) | Si lega al DNA a singolo filamento per impedirne il riavvolgimento. |
Collegamento all'apprendimento
[modifica | modifica sorgente]Per conoscere l'intero processo di replicazione del DNA guarda questo video su YouTube: DNA Replication - Oxford Academic (Oxford University Press).
Replicazione del DNA negli eucarioti
[modifica | modifica sorgente]I genomi eucariotici sono molto più complessi e di dimensioni maggiori rispetto ai genomi procariotici. Gli eucarioti hanno anche un certo numero di cromosomi lineari diversi. Il genoma umano ha 3 miliardi di coppie di basi per set aploide di cromosomi e 6 miliardi di coppie di basi vengono replicate durante la fase S del ciclo cellulare. Ci sono molteplici origini di replicazione su ogni cromosoma eucariotico; gli esseri umani possono avere fino a 100.000 origini di replicazione attraverso il genoma. La velocità di replicazione è di circa 100 nucleotidi al secondo, molto più lenta della replicazione procariotica. Nel lievito, che è un eucariote, sui cromosomi si trovano sequenze speciali note come sequenze a replica autonoma (ARS). Queste sono equivalenti all'origine di replicazione in E. coli .
Il numero di DNA polimerasi negli eucarioti è molto più alto che nei procarioti: ne sono note 14, di cui cinque sono note per avere ruoli importanti durante la replicazione e sono state ampiamente studiate. Sono note come pol α , pol β , pol γ , pol δ e pol ε .
I passaggi essenziali della replicazione sono gli stessi dei procarioti. Prima che la replicazione possa iniziare, il DNA deve essere reso disponibile come stampo. Il DNA eucariotico è legato a proteine basiche note come istoni per formare strutture chiamate nucleosomi. Gli istoni devono essere rimossi e poi sostituiti durante il processo di replicazione, il che aiuta a spiegare il tasso di replicazione inferiore negli eucarioti. La cromatina (il complesso tra DNA e proteine) può subire alcune modifiche chimiche, in modo che il DNA possa essere in grado di scivolare via dalle proteine o essere accessibile agli enzimi del macchinario di replicazione del DNA. All'origine della replicazione, un complesso di pre-replicazione viene creato con altre proteine iniziatrici. L'elicasi e altre proteine vengono quindi reclutate per avviare il processo di replicazione ( Tabella sotto).
Differenza tra replicazione procariotica ed eucariotica
| Proprietà | Procarioti | Eucarioti |
|---|---|---|
| Origine della replicazione | Separare | Multiplo |
| Tasso di replicazione | 1000 nucleotidi/s | Da 50 a 100 nucleotidi/s |
| Tipi di DNA polimerasi | 5 | 14 |
| Telomerasi | Non presente | Presente |
| Rimozione del primer RNA | DNA pol. I | RNasi H |
| Allungamento del filamento | DNA pol III | Pol a, pol d, pol e |
| Morsetto scorrevole | Morsetto scorrevole | PCNA |
Un'elicasi che utilizza l'energia dell'idrolisi dell'ATP apre l'elica del DNA. Le forcelle di replicazione si formano a ogni origine di replicazione mentre il DNA si svolge. L'apertura della doppia elica provoca un avvolgimento eccessivo, o superavvolgimento, nel DNA prima della forcella di replicazione. Questi vengono risolti con l'azione delle topoisomerasi. I primer sono formati dall'enzima primasi e, utilizzando il primer, la DNA pol può iniziare la sintesi. Sono quindi coinvolte tre principali DNA polimerasi: α, δ ed ε. La DNA pol α aggiunge un frammento di DNA corto (da 20 a 30 nucleotidi) al primer dell'RNA su entrambi i filamenti e poi passa a una seconda polimerasi. Mentre il filamento principale è sintetizzato continuamente dall'enzima pol ε , il filamento in ritardo è sintetizzato dalla pol δ . Una proteina a morsetto scorrevole nota come PCNA (antigene nucleare delle cellule proliferanti) mantiene la DNA pol in posizione in modo che non scivoli via dal DNA. Quando la pol δ si imbatte nell'RNA primer sul filamento in ritardo, lo sposta dal modello di DNA. L'RNA primer spostato viene quindi rimosso dalla RNasi H (nota anche come endonucleasi flap ) e sostituito con nucleotidi di DNA. I frammenti di Okazaki nel filamento in ritardo vengono uniti dopo la sostituzione dei primer di RNA con DNA. Gli spazi vuoti che rimangono vengono sigillati dalla DNA ligasi, che forma il legame fosfodiesterico.
Replicazione dei telomeri
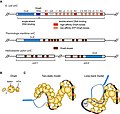
[modifica | modifica sorgente]A differenza dei cromosomi procariotici, i cromosomi eucariotici sono lineari. Come hai imparato, l'enzima DNA pol può aggiungere nucleotidi solo nella direzione 5'-3'. Nel filamento principale, la sintesi continua fino a quando non viene raggiunta la fine del cromosoma. Sul filamento ritardato, il DNA viene sintetizzato in brevi tratti, ognuno dei quali è iniziato da un primer separato. Quando la forcella di replicazione raggiunge la fine del cromosoma lineare, non c'è modo di sostituire il primer all'estremità 5' del filamento ritardato. Il DNA alle estremità del cromosoma rimane quindi spaiato e, nel tempo, queste estremità, chiamate telomeri, possono accorciarsi progressivamente man mano che le cellule continuano a dividersi.
I telomeri comprendono sequenze ripetitive che non codificano alcun gene in particolare. Negli esseri umani, una sequenza di sei coppie di basi, TTAGGG, viene ripetuta da 100 a 1000 volte nelle regioni dei telomeri. In un certo senso, questi telomeri proteggono i geni dall'eliminazione mentre le cellule continuano a dividersi. I telomeri vengono aggiunti alle estremità dei cromosomi da un enzima separato, la telomerasi ( Figura sotto), la cui scoperta ha aiutato a comprendere come vengono mantenute queste estremità ripetitive dei cromosomi. L' enzima telomerasi contiene una parte catalitica e un modello di RNA incorporato. Si attacca all'estremità del cromosoma e i nucleotidi di DNA complementari al modello di RNA vengono aggiunti all'estremità 3' del filamento di DNA. Una volta che l'estremità 3' del modello del filamento in ritardo è sufficientemente allungata, la DNA polimerasi può aggiungere i nucleotidi complementari alle estremità dei cromosomi. Pertanto, le estremità dei cromosomi vengono replicate.

La telomerasi è tipicamente attiva nelle cellule germinali e nelle cellule staminali adulte. Non è attiva nelle cellule somatiche adulte. Per la loro scoperta della telomerasi e della sua azione, Elizabeth Blackburn, Carol W. Greider e Jack W. Szostak hanno ricevuto il Premio Nobel per la Medicina e la Fisiologia nel 2009. Ricerche successive condotte utilizzando cellule HeLa (ottenute da Henrietta Lacks) hanno confermato che la telomerasi è presente nelle cellule umane. E nel 2001, ricercatori tra cui Diane L. Wright hanno scoperto che la telomerasi è necessaria per la rapida proliferazione delle cellule negli embrioni umani.
Telomerasi e invecchiamento
[modifica | modifica sorgente]Le cellule che subiscono la divisione cellulare continuano ad avere i loro telomeri accorciati perché la maggior parte delle cellule somatiche non produce telomerasi. Ciò significa essenzialmente che l'accorciamento dei telomeri è associato all'invecchiamento. Con l'avvento della medicina moderna, dell'assistenza sanitaria preventiva e di stili di vita più sani, la durata della vita umana è aumentata e c'è una crescente richiesta di persone che sembrano più giovani e hanno una migliore qualità della vita man mano che invecchiano.
Nel 2010, gli scienziati hanno scoperto che la telomerasi può invertire alcune condizioni legate all'età nei topi. Ciò potrebbe avere un potenziale nella medicina rigenerativa. 2 In questi studi sono stati utilizzati topi carenti di telomerasi; questi topi presentano atrofia tissutale, deplezione delle cellule staminali, insufficienza del sistema di organi e risposte compromesse alle lesioni tissutali. La riattivazione della telomerasi in questi topi ha causato l'estensione dei telomeri, ridotto il danno al DNA, invertito la neurodegenerazione e migliorato la funzione dei testicoli, della milza e dell'intestino. Pertanto, la riattivazione dei telomeri potrebbe avere un potenziale per il trattamento delle malattie legate all'età negli esseri umani.
Il cancro è caratterizzato dalla divisione cellulare incontrollata di cellule anomale. Le cellule accumulano mutazioni, proliferano in modo incontrollato e possono migrare in diverse parti del corpo attraverso un processo chiamato metastasi. Gli scienziati hanno osservato che le cellule cancerose hanno telomeri notevolmente accorciati e che la telomerasi è attiva in queste cellule. È interessante notare che solo dopo che i telomeri sono stati accorciati nelle cellule cancerose la telomerasi è diventata attiva. Se l'azione della telomerasi in queste cellule può essere inibita dai farmaci durante la terapia del cancro, allora le cellule cancerose potrebbero potenzialmente essere fermate dall'ulteriore divisione.
La riparazione del DNA
[modifica | modifica sorgente]La replicazione del DNA è un processo altamente accurato, ma occasionalmente possono verificarsi degli errori, come l'inserimento di una base sbagliata da parte di una DNA polimerasi. Errori non corretti possono talvolta portare a gravi conseguenze, come il cancro. I meccanismi di riparazione correggono gli errori. In rari casi, gli errori non vengono corretti, portando a mutazioni; in altri casi, gli enzimi di riparazione sono essi stessi mutati o difettosi.
La maggior parte degli errori durante la replicazione del DNA vengono prontamente corretti dalla capacità di correzione di bozze della DNA polimerasi stessa. ( Figura sotto). Nella correzione di bozze (proofreading) , la DNA pol legge la base appena aggiunta prima di aggiungere quella successiva, così da poter apportare una correzione. La polimerasi controlla se la base appena aggiunta si è accoppiata correttamente con la base nel filamento stampo. Se è la base giusta, viene aggiunto il nucleotide successivo. Se è stata aggiunta una base errata, l'enzima effettua un taglio al legame fosfodiesterico e rilascia il nucleotide sbagliato. Ciò viene eseguito dall'azione esonucleasica 3' della DNA pol. Una volta rimosso il nucleotide errato, può essere sostituito da quello corretto.

Alcuni errori non vengono corretti durante la replicazione, ma vengono invece corretti dopo che la replicazione è completata; questo tipo di riparazione è noto come riparazione di mismatch (anomalie di appaiamento) ( Figura sotto). Specifici enzimi di riparazione riconoscono il nucleotide non appaiato ed escisdono la parte del filamento che lo contiene; la regione escissa viene quindi risintetizzata. Se il mismatch non viene corretto, potrebbe causare danni più permanenti quando il DNA non appaiato viene replicato. In che modo gli enzimi di riparazione di mismatch riconoscono quale delle due basi è quella errata? In E. coli , dopo la replicazione, la base azotata adenina acquisisce un gruppo metilico; il filamento di DNA parentale avrà gruppi metilici, mentre il filamento appena sintetizzato ne è privo. Pertanto, la DNA polimerasi è in grado di rimuovere le basi erroneamente incorporate dal filamento appena sintetizzato, non metilato. Negli eucarioti il meccanismo non è ben compreso, ma si ritiene che implichi il riconoscimento di intaccature non sigillate nel nuovo filamento, nonché un'associazione continuativa a breve termine di alcune delle proteine di replicazione con il nuovo filamento figlio dopo il completamento della replicazione.

Un altro tipo di meccanismo di riparazione, la riparazione per escissione di nucleotidi , è simile alla riparazione per mismatch, tranne per il fatto che viene utilizzata per rimuovere le basi danneggiate anziché quelle non abbinate. Gli enzimi di riparazione sostituiscono le basi anomale effettuando un taglio sia sulle estremità 3' che 5' della base danneggiata ( Figura 14.19 ). Il segmento di DNA viene rimosso e sostituito con i nucleotidi correttamente accoppiati dall'azione della DNA pol. Una volta riempite le basi, lo spazio rimanente viene sigillato con un legame fosfodiesterico catalizzato dalla DNA ligasi. Questo meccanismo di riparazione viene spesso impiegato quando l'esposizione ai raggi UV provoca la formazione di dimeri di pirimidina.

Un esempio ben studiato di errori non corretti si riscontra nelle persone affette da xeroderma pigmentosa ( Figura sotto). Gli individui affetti hanno una pelle altamente sensibile ai raggi UV del sole. Quando gli individui sono esposti alla luce UV, si formano dimeri di pirimidina, in particolare quelli di timina; le persone affette da xeroderma pigmentosa non sono in grado di riparare il danno. Questi non vengono riparati a causa di un difetto negli enzimi di riparazione dell'escissione dei nucleotidi, mentre negli individui normali, i dimeri di timina vengono escissi e il difetto viene corretto. I dimeri di timina distorcono la struttura della doppia elica del DNA e questo può causare problemi durante la replicazione del DNA. Le persone affette da xeroderma pigmentosa possono avere un rischio maggiore di contrarre il cancro della pelle rispetto a coloro che non hanno la condizione.

Gli errori durante la replicazione del DNA non sono l'unica ragione per cui si verificano mutazioni nel DNA. Le mutazioni , variazioni nella sequenza nucleotidica di un genoma, possono verificarsi anche a causa di danni al DNA. Tali mutazioni possono essere di due tipi: indotte o spontanee. Le mutazioni indotte sono quelle che derivano da un'esposizione a sostanze chimiche, raggi UV, raggi X o qualche altro agente ambientale. Ad esempio, Charlotte Auerbach e JM Robson hanno scoperto gli effetti di induzione delle mutazioni del gas mostarda. Le mutazioni spontanee si verificano senza alcuna esposizione a nessun agente ambientale; sono il risultato di reazioni naturali che hanno luogo all'interno del corpo.
Le mutazioni possono avere un'ampia gamma di effetti. Le mutazioni puntiformi sono quelle mutazioni che interessano una singola coppia di basi. Le mutazioni nucleotidiche più comuni sono le sostituzioni, in cui una base viene sostituita da un'altra. Queste sostituzioni possono essere di due tipi, transizioni o trasversioni. La sostituzione di transizione si riferisce alla sostituzione di una purina o pirimidina con una base dello stesso tipo; ad esempio, una purina come l'adenina può essere sostituita dalla purina guanina. La sostituzione di trasversione si riferisce alla sostituzione di una purina con una pirimidina, o viceversa; ad esempio, la citosina, una pirimidina, viene sostituita dall'adenina, una purina. Alcune mutazioni puntiformi non sono rilevabili nel prodotto finale; queste sono note come mutazioni silenti. Le mutazioni silenti sono solitamente dovute a una sostituzione nella terza base di un codone, che spesso rappresenta lo stesso amminoacido del codone originale. Altre mutazioni puntiformi possono causare la sostituzione di un amminoacido con un altro, il che può alterare la funzione della proteina. Le mutazioni puntiformi che generano un codone di stop possono interrompere precocemente la produzione di una proteina.
Alcune mutazioni possono determinare un numero maggiore di copie dello stesso codone. Queste sono chiamate espansioni di ripetizione trinucleotidica e determinano regioni ripetute dello stesso amminoacido. Le mutazioni possono anche essere il risultato dell'aggiunta di una base, nota come inserzione, o della rimozione di una base, nota anche come delezione. Se un'inserzione o una delezione determina l'alterazione del frame di lettura traslazionale (una mutazione frameshift), la proteina risultante è solitamente non funzionale. A volte un pezzo di DNA da un cromosoma può essere traslocato in un altro cromosoma o in un'altra regione dello stesso cromosoma; questo è anche noto come traslocazione. Questi tipi di mutazione sono mostrati nella Figura sotto.

Esercizio: una mutazione frameshift che determina l'inserimento di tre nucleotidi è spesso meno deleteria di una mutazione che determina l'inserimento di un nucleotide. Perché?
È noto che le mutazioni nei geni di riparazione causano il cancro. Molti geni di riparazione mutati sono stati implicati in alcune forme di cancro al pancreas, cancro al colon e cancro colorettale. Le mutazioni possono colpire sia le cellule somatiche che le cellule germinali. Se molte mutazioni si accumulano in una cellula somatica, possono portare a problemi come la divisione cellulare incontrollata osservata nel cancro. Se una mutazione avviene nelle cellule germinali, la mutazione verrà trasmessa alla generazione successiva, come nel caso dell'emofilia e della xeroderma pigmentosa.