Biologia per il liceo/La sintesi proteica
Dalla riscoperta del lavoro di Mendel nel 1900, la definizione del gene è passata da un'unità astratta di ereditarietà a un'entità molecolare tangibile capace di replicazione, espressione e mutazione ( Figura 15.1 ). I geni sono composti da DNA e sono disposti linearmente sui cromosomi. I geni specificano le sequenze di amminoacidi, che sono i mattoni delle proteine. A loro volta, le proteine sono responsabili dell'orchestrazione di quasi ogni funzione della cellula. Sia i geni che le proteine che codificano sono assolutamente essenziali per la vita come la conosciamo.
Il codice genetico
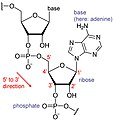
[modifica | modifica sorgente]Il processo cellulare di trascrizione genera RNA messaggero (mRNA), una copia molecolare mobile di uno o più geni con un alfabeto di A, C, G e uracile (U). La traduzione del modello di mRNA sui ribosomi converte le informazioni genetiche basate sui nucleotidi in un prodotto proteico. Questo è il dogma centrale della sintesi DNA-proteina. Le sequenze proteiche sono costituite da 20 amminoacidi comuni; pertanto, si può dire che l'alfabeto proteico è costituito da 20 "lettere" ( Figura sotto). Diversi amminoacidi hanno diverse chimiche (come acido contro basico, o polare e non polare) e diversi vincoli strutturali. La variazione nella sequenza di amminoacidi è responsabile dell'enorme variazione nella struttura e nella funzione delle proteine.

Il dogma centrale: il DNA codifica l'RNA; l'RNA codifica le proteine
[modifica | modifica sorgente]Il flusso di informazioni genetiche nelle cellule dal DNA all'mRNA alle proteine è descritto dal dogma centrale ( Figura sotto), che afferma che i geni specificano la sequenza degli mRNA, che a loro volta specificano la sequenza degli amminoacidi che compongono tutte le proteine. La decodifica di una molecola in un'altra è eseguita da proteine e RNA specifici. Poiché le informazioni immagazzinate nel DNA sono così centrali per la funzione cellulare, è intuitivo che la cellula faccia copie di mRNA di queste informazioni per la sintesi proteica, mantenendo il DNA stesso intatto e protetto. La copia del DNA in RNA è relativamente semplice, con un nucleotide aggiunto al filamento di mRNA per ogni nucleotide letto nel filamento di DNA. La traduzione in proteina è un po' più complessa perché tre nucleotidi di mRNA corrispondono a un amminoacido nella sequenza polipeptidica. Tuttavia, la traduzione in proteina è ancora sistematica e collineare , in modo tale che i nucleotidi da 1 a 3 corrispondano all'amminoacido 1, i nucleotidi da 4 a 6 corrispondano all'amminoacido 2 e così via.

Il codice genetico è degenerato e universale
[modifica | modifica sorgente]Ogni amminoacido è definito da una sequenza di tre nucleotidi chiamata codone di tripletta. Dati i diversi numeri di "lettere" negli "alfabeti" dell'mRNA e delle proteine, gli scienziati hanno teorizzato che i singoli amminoacidi debbano essere rappresentati da combinazioni di nucleotidi. Le doppiette di nucleotidi non sarebbero sufficienti a specificare ogni amminoacido perché ci sono solo 16 possibili combinazioni di due nucleotidi (4 2 ). Al contrario, ci sono 64 possibili triplette di nucleotidi (43), che sono molto di più del numero di amminoacidi. Gli scienziati hanno teorizzato che gli amminoacidi fossero codificati da triplette di nucleotidi e che il codice genetico fosse " degenerato ". In altre parole, un dato amminoacido potrebbe essere codificato da più di una tripletta di nucleotidi. Ciò è stato successivamente confermato sperimentalmente: Francis Crick e Sydney Brenner hanno utilizzato il mutageno chimico proflavina per inserire uno, due o tre nucleotidi nel gene di un virus. Quando venivano inseriti uno o due nucleotidi, le proteine normali non venivano prodotte. Quando venivano inseriti tre nucleotidi, la proteina veniva sintetizzata e resa funzionale. Ciò dimostrava che gli amminoacidi devono essere specificati da gruppi di tre nucleotidi. Queste triplette di nucleotidi sono chiamate codoni . L'inserimento di uno o due nucleotidi modificava completamente il frame di lettura della tripletta , alterando così il messaggio per ogni amminoacido successivo ( Figura 15.5 ). Sebbene l'inserimento di tre nucleotidi causasse l'inserimento di un amminoacido extra durante la traduzione, l'integrità del resto della proteina veniva mantenuta.
Gli scienziati hanno risolto con grande impegno il codice genetico traducendo mRNA sintetici in vitro e sequenziando le proteine da loro specificate ( Figura sotto).

Oltre ai codoni che istruiscono l'aggiunta di uno specifico amminoacido a una catena polipeptidica, tre dei 64 codoni terminano la sintesi proteica e rilasciano il polipeptide dal macchinario di traduzione. Queste triplette sono chiamate codoni nonsenso o codoni di stop . Un altro codone, AUG, ha anche una funzione speciale. Oltre a specificare l'amminoacido metionina, funge anche da codone di inizio per avviare la traduzione. Il frame di lettura per la traduzione è impostato dal codone di inizio AUG vicino all'estremità 5' dell'mRNA. Dopo il codone di inizio, l'mRNA viene letto in gruppi di tre fino a quando non viene incontrato un codone di stop.
La disposizione della tabella di codifica rivela la struttura del codice. Ci sono sedici "blocchi" di codoni, ciascuno specificato dal primo e dal secondo nucleotide dei codoni all'interno del blocco, ad esempio, il blocco "AC*" che corrisponde all'amminoacido treonina (Thr). Alcuni blocchi sono divisi in una metà pirimidinica, in cui il codone termina con U o C, e una metà purinica, in cui il codone termina con A o G. Alcuni amminoacidi ottengono un intero blocco di quattro codoni, come alanina (Ala), treonina (Thr) e prolina (Pro). Alcuni ottengono la metà pirimidinica del loro blocco, come istidina (His) e asparagina (Asn). Altri ottengono la metà purinica del loro blocco, come glutammato (Glu) e lisina (Lys). Nota che alcuni amminoacidi ottengono un blocco e mezzo blocco per un totale di sei codoni.
La specificazione di un singolo amminoacido da parte di più codoni simili è chiamata "degenerazione". Si ritiene che la degenerazione sia un meccanismo cellulare per ridurre l'impatto negativo delle mutazioni casuali. I codoni che specificano lo stesso amminoacido in genere differiscono solo di un nucleotide. Inoltre, gli amminoacidi con catene laterali chimicamente simili sono codificati da codoni simili. Ad esempio, l'aspartato (Asp) e il glutammato (Glu), che occupano il blocco GA*, sono entrambi caricati negativamente. Questa sfumatura del codice genetico assicura che una mutazione di sostituzione di un singolo nucleotide possa specificare lo stesso amminoacido ma non avere alcun effetto o specificare un amminoacido simile, impedendo alla proteina di essere resa completamente non funzionale.
Il codice genetico è quasi universale. Con poche eccezioni minori, praticamente tutte le specie usano lo stesso codice genetico per la sintesi proteica. La conservazione dei codoni significa che un mRNA purificato che codifica la proteina globina nei cavalli potrebbe essere trasferito a una cellula di tulipano, e il tulipano sintetizzerebbe la globina di cavallo. Il fatto che ci sia un solo codice genetico è una prova schiacciante che tutta la vita sulla Terra condivide un'origine comune, soprattutto considerando che ci sono circa 10 84 possibili combinazioni di 20 amminoacidi e 64 codoni tripletti.
Collegamento all'apprendimento
[modifica | modifica sorgente]Trascrivere un gene e tradurlo in proteina utilizzando l'appaiamento complementare e il codice genetico in questo sito.

| Laboratorio |
|---|
Chi ha più DNA: un kiwi o una fragola?[modifica | modifica sorgente]Pensi che un kiwi o una fragola abbiano più DNA per frutto? Domanda : Un kiwi e una fragola che hanno all'incirca le stesse dimensioni ( Figura 15.6 ) avrebbero all'incirca la stessa quantità di DNA? Contesto : i geni sono trasportati sui cromosomi e sono fatti di DNA. Tutti i mammiferi sono diploidi, ovvero hanno due copie di ogni cromosoma. Tuttavia, non tutte le piante sono diploidi. La fragola comune è ottoploide (8 n ) e il kiwi coltivato è esaploide (6 n ). Fai una ricerca sul numero totale di cromosomi nelle cellule di ciascuno di questi frutti e pensa a come questo potrebbe corrispondere alla quantità di DNA nei nuclei cellulari di questi frutti. Quali altri fattori potrebbero contribuire alla quantità totale di DNA in un singolo frutto? Leggi la tecnica di isolamento del DNA per capire come ogni passaggio del protocollo di isolamento aiuta a liberare e precipitare il DNA. Ipotesi : ipotizza se saresti in grado di rilevare una differenza nella quantità di DNA da fragole e kiwi di dimensioni simili. Quale frutto pensi che produrrebbe più DNA? Metti alla prova la tua ipotesi : isola il DNA da una fragola e da un kiwi di dimensioni simili. Esegui l'esperimento almeno in triplicato per ogni frutto
Registra le tue osservazioni : poiché non stai misurando quantitativamente il volume del DNA, puoi registrare per ogni prova se i due frutti hanno prodotto la stessa quantità di DNA o quantità diverse rispetto a quelle osservate a occhio nudo. Se uno o l'altro frutto ha prodotto notevolmente più DNA, registra anche questo. Determina se le tue osservazioni sono coerenti con diversi pezzi di ciascun frutto. Analizza i tuoi dati : hai notato una differenza evidente nella quantità di DNA prodotta da ogni frutto? I tuoi risultati erano riproducibili? Trai una conclusione : dato ciò che sai sul numero di cromosomi in ogni frutto, puoi concludere che il numero di cromosomi è necessariamente correlato alla quantità di DNA? Puoi identificare eventuali svantaggi di questa procedura? Se avessi accesso a un laboratorio, come potresti standardizzare il tuo confronto e renderlo più quantitativo? |
La trascrizione nei procarioti
[modifica | modifica sorgente]I procarioti, che includono batteri e archea, sono per lo più organismi unicellulari che, per definizione, sono privi di nuclei legati alla membrana e di altri organelli. Un cromosoma batterico è un cerchio chiuso che, a differenza dei cromosomi eucariotici, non è organizzato attorno alle proteine istoniche. La regione centrale della cellula in cui risiede il DNA procariotico è chiamata regione nucleoide. Inoltre, i procarioti hanno spesso abbondanti plasmidi , che sono molecole di DNA più corte e circolari che possono contenere solo uno o pochi geni. I plasmidi possono essere trasferiti indipendentemente dal cromosoma batterico durante la divisione cellulare e spesso portano tratti come quelli coinvolti nella resistenza agli antibiotici.
La trascrizione nei procarioti (e negli eucarioti) richiede che la doppia elica del DNA si srotoli parzialmente nella regione di sintesi dell'mRNA. La regione di srotolamento è chiamata bolla di trascrizione. La trascrizione procede sempre dallo stesso filamento di DNA per ogni gene, che è chiamato filamento stampo . Il prodotto dell'mRNA è complementare al filamento stampo ed è quasi identico all'altro filamento di DNA, chiamato filamento non stampo o filamento codificante. L'unica differenza nucleotidica è che nell'mRNA tutti i nucleotidi T sono sostituiti con nucleotidi U ( Figura sotto). In una doppia elica di RNA, A può legare U tramite due legami idrogeno, proprio come nell'appaiamento A–T in una doppia elica di DNA.

La coppia di nucleotidi nella doppia elica del DNA che corrisponde al sito da cui viene trascritto il primo nucleotide 5' mRNA è chiamata sito +1 o sito di inizio . I nucleotidi che precedono il sito di inizio sono indicati con un "-" e sono designati nucleotidi a monte . Al contrario, i nucleotidi che seguono il sito di inizio sono indicati con la numerazione "+" e sono chiamati nucleotidi a valle .
Inizio della trascrizione nei procarioti
[modifica | modifica sorgente]I procarioti non hanno nuclei racchiusi da membrana. Pertanto, i processi di trascrizione, traduzione e degradazione dell'mRNA possono verificarsi tutti simultaneamente. Il livello intracellulare di una proteina batterica può essere rapidamente amplificato da più eventi di trascrizione e traduzione che si verificano contemporaneamente sullo stesso modello di DNA. I genomi procariotici sono molto compatti e le trascrizioni procariotiche spesso coprono più di un gene o cistrone (una sequenza codificante per una singola proteina). Gli mRNA policistronici vengono quindi tradotti per produrre più di un tipo di proteina.
La nostra discussione qui esemplificherà la trascrizione descrivendo questo processo in Escherichia coli , una specie eubatterica ben studiata. Sebbene esistano alcune differenze tra la trascrizione in E. coli e la trascrizione negli archaea, una comprensione della trascrizione di E. coli può essere applicata a praticamente tutte le specie batteriche.
RNA polimerasi procariotica
[modifica | modifica sorgente]I procarioti usano la stessa RNA polimerasi per trascrivere tutti i loro geni. In E. coli , la polimerasi è composta da cinque subunità polipeptidiche, due delle quali sono identiche. Quattro di queste subunità, denotate α , α , β e β ', comprendono l' enzima core della polimerasi . Queste subunità si assemblano ogni volta che un gene viene trascritto e si disassemblano una volta completata la trascrizione. Ogni subunità ha un ruolo unico; le due subunità α sono necessarie per assemblare la polimerasi sul DNA; la subunità β si lega al ribonucleoside trifosfato che diventerà parte della molecola di mRNA nascente; e la subunità β ' si lega al filamento stampo del DNA. La quinta subunità, σ , è coinvolta solo nell'inizio della trascrizione. Conferisce specificità trascrizionale tale che la polimerasi inizia a sintetizzare mRNA da un sito di inizio appropriato. Senza σ , l'enzima centrale trascriverebbe da siti casuali e produrrebbe molecole di mRNA che specificano il linguaggio incomprensibile delle proteine. La polimerasi composta da tutte e cinque le subunità è chiamata oloenzima .
Promotori procariotici
[modifica | modifica sorgente]Un promotore è una sequenza di DNA su cui il macchinario di trascrizione, inclusa la RNA polimerasi, si lega e avvia la trascrizione. Nella maggior parte dei casi, i promotori esistono a monte dei geni che regolano. La sequenza specifica di un promotore è molto importante perché determina se il gene corrispondente viene trascritto sempre, a volte o raramente. Sebbene i promotori varino tra i genomi procariotici, alcuni elementi sono conservati evolutivamente in molte specie. Nelle regioni -10 e -35 a monte del sito di inizio, ci sono due sequenze di consenso del promotore , o regioni che sono simili in tutti i promotori e in varie specie batteriche ( Figura 15.8 ). La sequenza -10, chiamata regione -10, ha la sequenza di consenso TATAAT. La sequenza -35 ha la sequenza di consenso TTGACA. Queste sequenze di consenso sono riconosciute e legate da σ . Una volta che questa interazione è avvenuta, le subunità dell'enzima centrale si legano al sito. La regione -10 ricca di A–T facilita lo svolgimento del modello di DNA e vengono creati diversi legami fosfodiesterici. La fase di inizio della trascrizione termina con la produzione di trascrizioni abortive, che sono polimeri di circa 10 nucleotidi che vengono creati e rilasciati.

Collegamento all'apprendimento
[modifica | modifica sorgente]Guarda questa animazione su YouTube per vedere il processo di trascrizione mentre avviene nella cellula: Bacterial Transcription di Susan Howitt.
Allungamento e terminazione nei procarioti
[modifica | modifica sorgente]La fase di allungamento della trascrizione inizia con il rilascio della subunità σ dalla polimerasi. La dissociazione di σ consente all'enzima centrale di procedere lungo il modello di DNA, sintetizzando mRNA nella direzione 5'-3' a una velocità di circa 40 nucleotidi al secondo. Man mano che l'allungamento procede, il DNA viene continuamente srotolato davanti all'enzima centrale e riavvolto dietro di esso. L'appaiamento delle basi tra DNA e RNA non è abbastanza stabile da mantenere la stabilità dei componenti di sintesi dell'mRNA. Invece, l'RNA polimerasi agisce come un linker stabile tra il modello di DNA e i filamenti di RNA nascenti per garantire che l'allungamento non venga interrotto prematuramente.
Segnali di terminazione procariotici
[modifica | modifica sorgente]Una volta che un gene è stato trascritto, la polimerasi procariotica deve essere istruita a dissociarsi dal modello di DNA e liberare l'mRNA appena creato. A seconda del gene che viene trascritto, ci sono due tipi di segnali di terminazione. Uno è basato sulle proteine e l'altro è basato sull'RNA. La terminazione Rho-dipendente è controllata dalla proteina rho (ρ), che segue la polimerasi sulla catena di mRNA in crescita. Vicino alla fine del gene, la polimerasi incontra una serie di nucleotidi G sul modello di DNA e si blocca. Di conseguenza, la proteina rho collide con la polimerasi. L'interazione con rho rilascia l'mRNA dalla bolla di trascrizione.
La terminazione Rho-indipendente è controllata da sequenze specifiche nel filamento stampo del DNA. Quando la polimerasi si avvicina alla fine del gene che viene trascritto, incontra una regione ricca di nucleotidi C–G. L'mRNA si ripiega su se stesso e i nucleotidi complementari C–G si legano insieme. Il risultato è una forcina stabile che fa sì che la polimerasi si blocchi non appena inizia a trascrivere una regione ricca di nucleotidi A–T. La regione complementare U–A della trascrizione dell'mRNA forma solo un'interazione debole con il DNA stampo. Questo, insieme alla polimerasi bloccata, induce sufficiente instabilità affinché l'enzima centrale si stacchi e liberi la nuova trascrizione dell'mRNA.
Al termine, il processo di trascrizione è completo. Al momento della terminazione, la trascrizione procariotica sarebbe già stata utilizzata per iniziare la sintesi di numerose copie della proteina codificata perché questi processi possono verificarsi contemporaneamente. L'unificazione della trascrizione, della traduzione e persino della degradazione dell'mRNA è possibile perché tutti questi processi si verificano nella stessa direzione 5'-3' e perché non c'è alcuna compartimentazione membranosa nella cellula procariotica ( Figura 15.9 ). Al contrario, la presenza di un nucleo nelle cellule eucariotiche impedisce la trascrizione e la traduzione simultanee.

Collegamento all'apprendimento
[modifica | modifica sorgente]Visita questa animazione su YouTube per vedere il processo di trascrizione procariotica: Transcription di ndsuvirtualcell.
-
Operone Lac nei batteri
-
Operone lac
La trascrizione negli eucarioti
[modifica | modifica sorgente]I procarioti e gli eucarioti eseguono fondamentalmente lo stesso processo di trascrizione, con alcune differenze fondamentali. La differenza più importante tra la trascrizione dei procarioti e degli eucarioti è dovuta al nucleo e agli organelli legati alla membrana di quest'ultimi. Con i geni legati in un nucleo, la cellula eucariotica deve essere in grado di trasportare il suo mRNA nel citoplasma e deve proteggere il suo mRNA dalla degradazione prima che venga tradotto. Gli eucarioti impiegano anche tre diverse polimerasi che trascrivono ciascuna un diverso sottoinsieme di geni. Gli mRNA eucariotici sono solitamente monogenici , il che significa che specificano una singola proteina.
Inizio della trascrizione negli eucarioti
[modifica | modifica sorgente]A differenza della polimerasi procariotica che può legarsi autonomamente a uno stampo di DNA, gli eucarioti necessitano di diverse altre proteine, chiamate fattori di trascrizione, che si legano prima alla regione del promotore e poi aiutano a reclutare la polimerasi appropriata.

Le tre RNA polimerasi eucariotiche
[modifica | modifica sorgente]Le caratteristiche della sintesi di mRNA eucariotica sono notevolmente più complesse di quelle dei procarioti. Invece di una singola polimerasi composta da cinque subunità, gli eucarioti hanno tre polimerasi, ciascuna composta da 10 subunità o più. Ogni polimerasi eucariotica richiede anche un set distinto di fattori di trascrizione per portarla al modello di DNA.
L'RNA polimerasi I si trova nel nucleolo, una sottostruttura nucleare specializzata in cui l'RNA ribosomiale (rRNA) viene trascritto, elaborato e assemblato nei ribosomi ( Tabella sotto). Le molecole di rRNA sono considerate RNA strutturali perché hanno un ruolo cellulare ma non vengono tradotte in proteine. Gli rRNA sono componenti del ribosoma e sono essenziali per il processo di traduzione. L'RNA polimerasi I sintetizza tutti gli rRNA dal set duplicato in tandem di geni ribosomiali 18S, 5.8S e 28S. (Si noti che la designazione "S" si applica alle unità "Svedberg", un valore non additivo che caratterizza la velocità alla quale una particella sedimenta durante la centrifugazione.)
Posizioni, prodotti e sensibilità delle tre RNA polimerasi eucariotiche
| RNA polimerasi | Compartimento cellulare | Prodotto della trascrizione | Sensibilità all'α-Amanitina |
|---|---|---|---|
| IO | Nucleolo | Tutti gli rRNA tranne l'rRNA 5S | Insensibile |
| Io sono | Nucleo | Tutti i pre-mRNA nucleari codificanti proteine | Estremamente sensibile |
| III | Nucleo | 5S rRNA, tRNA e piccoli RNA nucleari | Moderatamente sensibile |
La RNA polimerasi II è localizzata nel nucleo e sintetizza tutti i pre-mRNA nucleari che codificano le proteine. I pre-mRNA eucariotici subiscono un'elaborazione estesa dopo la trascrizione ma prima della traduzione. Per chiarezza, la discussione di questo modulo sulla trascrizione e la traduzione negli eucarioti userà il termine "mRNA" per descrivere solo le molecole mature ed elaborate che sono pronte per essere tradotte. La RNA polimerasi II è responsabile della trascrizione della stragrande maggioranza dei geni eucariotici.
Anche la RNA polimerasi III si trova nel nucleo. Questa polimerasi trascrive una varietà di RNA strutturali che includono il pre-rRNA 5S, i pre-RNA di trasferimento (pre-tRNA) e i piccoli pre- RNA nucleari . I tRNA hanno un ruolo critico nella traduzione; fungono da "molecole adattatrici" tra il modello di mRNA e la catena polipeptidica in crescita. I piccoli RNA nucleari hanno una varietà di funzioni, tra cui lo "splicing" dei pre-mRNA e la regolazione dei fattori di trascrizione.
Uno scienziato che caratterizza un nuovo gene può determinare quale polimerasi lo trascrive testando se il gene è espresso in presenza di α-amanitina, una tossina oligopeptidica prodotta dal fungo agarico e da altre specie di Amanita . È interessante notare che l'α-amanitina influenza le tre polimerasi in modo molto diverso ( Tabella 15.1 ). La RNA polimerasi I è completamente insensibile all'α-amanitina, il che significa che la polimerasi può trascrivere il DNA in vitro in presenza di questo veleno. La RNA polimerasi III è moderatamente sensibile alla tossina. Al contrario, la RNA polimerasi II è estremamente sensibile all'α-amanitina. La tossina impedisce all'enzima di progredire lungo il DNA e quindi inibisce la trascrizione. Conoscere la polimerasi trascrizionale può fornire indizi sulla funzione generale del gene studiato. Poiché l'RNA polimerasi II trascrive la stragrande maggioranza dei geni, nelle nostre successive discussioni sui fattori di trascrizione e sui promotori eucariotici ci concentreremo su questa polimerasi.
Promotori dell'RNA polimerasi II e fattori di trascrizione
[modifica | modifica sorgente]I promotori eucariotici sono molto più grandi e intricati dei promotori procariotici. Tuttavia, entrambi hanno una sequenza simile alla sequenza -10 dei procarioti. Negli eucarioti, questa sequenza è chiamata TATA box e ha la sequenza di consenso TATAAA sul filamento codificante. Si trova a -25-35 basi rispetto al sito di inizio (+1) ( Figura sotto). Questa sequenza non è identica alla E. coli -10 box, ma conserva l'elemento ricco di A–T. La termostabilità dei legami A–T è bassa e questo aiuta lo stampo del DNA a svolgersi localmente in preparazione alla trascrizione.
Invece del semplice fattore σ che aiuta a legare la RNA polimerasi procariotica al suo promotore, gli eucarioti assemblano un complesso di fattori di trascrizione necessari per reclutare la RNA polimerasi II in un gene che codifica per proteine. I fattori di trascrizione che si legano al promotore sono chiamati fattori di trascrizione basali . Questi fattori basali sono tutti chiamati TFII (fattore di trascrizione/polimerasi II) più una lettera aggiuntiva (AJ). Il complesso centrale è TFIID, che include una proteina legante TATA (TBP). Gli altri fattori di trascrizione si agganciano al DNA, e ciascuno stabilizza ulteriormente il complesso di pre-inizio e contribuisce al reclutamento della RNA polimerasi II.

Alcuni promotori eucariotici hanno anche un box CAAT conservato (GGCCAATCT) a circa -80. Più a monte del box TATA, i promotori eucariotici possono anche contenere uno o più box ricchi di GC (GGCG) o box ottamerici (ATTTGCAT). Questi elementi legano fattori cellulari che aumentano l'efficienza dell'inizio della trascrizione e sono spesso identificati in geni più "attivi" che vengono costantemente espressi dalla cellula.
I fattori di trascrizione basali sono cruciali nella formazione di un complesso di pre-inizio sul modello di DNA che successivamente recluta la RNA polimerasi II per l'inizio della trascrizione. La complessità della trascrizione eucariotica non finisce con le polimerasi e i promotori. Un esercito di altri fattori di trascrizione, che si legano agli enhancer e ai silencer a monte, aiutano anche a regolare la frequenza con cui il pre-mRNA viene sintetizzato da un gene. Enhancer e silencer influenzano l'efficienza della trascrizione ma non sono necessari affinché la trascrizione proceda.
Strutture promotrici per RNA polimerasi I e III
[modifica | modifica sorgente]I processi di trasporto delle RNA polimerasi I e III allo stampo di DNA coinvolgono raccolte di fattori di trascrizione leggermente meno complesse, ma il tema generale è lo stesso.
Gli elementi promotori conservati per i geni trascritti dalle polimerasi I e III differiscono da quelli trascritti dalla RNA polimerasi II. La RNA polimerasi I trascrive geni che hanno due sequenze promotrici ricche di GC nella regione da -45 a +20. Queste sequenze da sole sono sufficienti per l'inizio della trascrizione, ma i promotori con sequenze aggiuntive nella regione da -180 a -105 a monte del sito di inizio miglioreranno ulteriormente l'inizio. I geni che sono trascritti dalla RNA polimerasi III hanno promotori a monte o promotori che si verificano all'interno dei geni stessi.
La trascrizione eucariotica è un processo strettamente regolato che richiede che una varietà di proteine interagiscano tra loro e con il filamento di DNA. Sebbene il processo di trascrizione negli eucarioti comporti un maggiore investimento metabolico rispetto ai procarioti, assicura che la cellula trascriva precisamente i pre-mRNA di cui ha bisogno per la sintesi proteica.
-
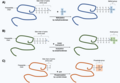
Gli elementi della sequenza regolatrice (giallo) all'inizio di un gene eucariotico che codifica per una proteina possono essere immediatamente a monte dell'open read frame (ORF, rosso) o a molte kilobasi di distanza (a monte o a valle). Le regioni promotore ed enhancer regolano positivamente (e i silenziatori regolano negativamente) la trascrizione dal DNA all'mRNA. Le regioni non tradotte 5' e 3' di quell'mRNA (UTR, blu) regolano quindi la traduzione nel prodotto proteico finale.
-
Durante l'inizio della trascrizione, le proteine (semicerchi grigio scuro) legate a regioni distanti del DNA possono essere avvicinate tra loro poiché il DNA intermedio può ripiegarsi su se stesso. Il macchinario di trascrizione basale può interagire con attivatori e repressori distanti molte kilobasi a monte o a valle dell'open reading frame.
-
Struttura della RNA polimerasi II complessata con il DNA modello (verde). Il filamento di RNA trascritto è in giallo.
-
il ruolo dei fattori di trascrizione
Allungamento e terminazione eucariotica
[modifica | modifica sorgente]Dopo la formazione del complesso di pre-inizio, la polimerasi viene rilasciata dagli altri fattori di trascrizione e l'allungamento può procedere come avviene nei procarioti con la polimerasi che sintetizza il pre-mRNA nella direzione 5'-3'. Come discusso in precedenza, la RNA polimerasi II trascrive la quota maggiore dei geni eucariotici, quindi in questa sezione ci concentreremo su come questa polimerasi realizza l'allungamento e la terminazione.

Sebbene il processo enzimatico di allungamento sia essenzialmente lo stesso negli eucarioti e nei procarioti, il modello del DNA è considerevolmente più complesso. Quando le cellule eucariotiche non si dividono, i loro geni esistono come una massa diffusa di DNA e proteine chiamata cromatina. Il DNA è strettamente impacchettato attorno a proteine istoniche cariche a intervalli ripetuti. Questi complessi DNA-istone , chiamati collettivamente nucleosomi, sono regolarmente distanziati e includono 146 nucleotidi di DNA avvolti attorno a otto istoni come un filo attorno a una bobina.
Per far sì che avvenga la sintesi dei polinucleotidi, il macchinario di trascrizione deve spostare gli istoni ogni volta che incontra un nucleosoma. Ciò è possibile grazie a uno speciale complesso proteico chiamato FACT , che sta per " facilita la trascrizione della cromatina ". Questo complesso allontana gli istoni dal modello di DNA mentre la polimerasi si muove lungo di esso. Una volta sintetizzato il pre-mRNA, il complesso FACT sostituisce gli istoni per ricreare i nucleosomi.
La terminazione della trascrizione è diversa per le diverse polimerasi. A differenza dei procarioti, l'allungamento da parte della RNA polimerasi II negli eucarioti avviene da 1.000 a 2.000 nucleotidi oltre la fine del gene che viene trascritto. Questa coda pre-mRNA viene successivamente rimossa tramite scissione durante l'elaborazione dell'mRNA. D'altro canto, le RNA polimerasi I e III richiedono segnali di terminazione. I geni trascritti dalla RNA polimerasi I contengono una sequenza specifica di 18 nucleotidi che viene riconosciuta da una proteina di terminazione. Il processo di terminazione nella RNA polimerasi III coinvolge una forcina per mRNA simile alla terminazione della trascrizione indipendente da rho nei procarioti.

L'elaborazione dell'mRNA negli eucarioti
[modifica | modifica sorgente]Dopo la trascrizione, i pre-mRNA eucariotici devono subire diverse fasi di elaborazione prima di poter essere tradotti. Anche i tRNA e gli rRNA eucariotici (e procariotici) subiscono un'elaborazione prima di poter funzionare come componenti nel macchinario di sintesi proteica.
Il pre-mRNA eucariotico subisce un'elaborazione estesa prima di essere pronto per essere tradotto. Le sequenze codificanti le proteine eucariotiche non sono continue, come nei procarioti. Le sequenze codificanti (esoni) sono interrotte da introni non codificanti, che devono essere rimossi per creare un mRNA traducibile. Gli ulteriori passaggi coinvolti nella maturazione dell'mRNA eucariotico creano anche una molecola con un'emivita molto più lunga di un mRNA procariotico. Gli mRNA eucariotici durano diverse ore, mentre il tipico mRNA di E. coli non dura più di cinque secondi.
I pre-mRNA vengono prima rivestiti con proteine stabilizzanti l'RNA; queste proteggono il pre-mRNA dalla degradazione mentre viene elaborato ed esportato fuori dal nucleo. I tre passaggi più importanti dell'elaborazione del pre-mRNA sono l'aggiunta di fattori stabilizzanti e di segnalazione alle estremità 5' e 3' della molecola e la rimozione degli introni ( Figura 15.11 ). In rari casi, la trascrizione dell'mRNA può essere "modificata" dopo essere stata trascritta.

5' capping
[modifica | modifica sorgente]Mentre il pre-mRNA è ancora in fase di sintesi, un cappuccio di 7-metilguanosina viene aggiunto all'estremità 5' del trascritto in crescita tramite un legame fosfato. Questo gruppo funzionale protegge l'mRNA nascente dalla degradazione. Inoltre, i fattori coinvolti nella sintesi proteica riconoscono il cappuccio per aiutare ad avviare la traduzione da parte dei ribosomi.
Coda in poli-A da 3'
[modifica | modifica sorgente]Una volta completato l'allungamento, il pre-mRNA viene scisso da un'endonucleasi tra una sequenza di consenso AAUAAA e una sequenza ricca di GU, lasciando la sequenza AAUAAA sul pre-mRNA. Un enzima chiamato poli-A polimerasi aggiunge quindi una stringa di circa 200 residui A, chiamata coda di poli-A . Questa modifica protegge ulteriormente il pre-mRNA dalla degradazione ed è anche il sito di legame per una proteina necessaria per esportare l'mRNA elaborato nel citoplasma.
Splicing pre-mRNA
[modifica | modifica sorgente]I geni eucariotici sono composti da esoni , che corrispondono a sequenze codificanti proteine ( es- on significa che sono espresse ), e sequenze intervenienti chiamate introni ( int- ron denota il loro ruolo interveniente ), che possono essere coinvolte nella regolazione genica ma vengono rimosse dal pre-mRNA durante l'elaborazione. Le sequenze introniche nell'mRNA non codificano proteine funzionali.
La scoperta degli introni fu una sorpresa per i ricercatori degli anni '70 che si aspettavano che i pre-mRNA avrebbero specificato sequenze proteiche senza ulteriore elaborazione, come avevano osservato nei procarioti. I geni degli eucarioti superiori contengono molto spesso uno o più introni. Queste regioni possono corrispondere a sequenze regolatrici; tuttavia, il significato biologico di avere molti introni o di avere introni molto lunghi in un gene non è chiaro. È possibile che gli introni rallentino l'espressione genica perché ci vuole più tempo per trascrivere i pre-mRNA con molti introni. In alternativa, gli introni potrebbero essere resti di sequenze non funzionali lasciati dalla fusione di geni antichi nel corso dell'evoluzione. Ciò è supportato dal fatto che esoni separati spesso codificano subunità o domini proteici separati. Per la maggior parte, le sequenze di introni possono essere mutate senza influenzare in ultima analisi il prodotto proteico.
Tutti gli introni di un pre-mRNA devono essere rimossi completamente e precisamente prima della sintesi proteica. Se il processo sbaglia anche di un solo nucleotide, il frame di lettura degli esoni riuniti si sposterebbe e la proteina risultante sarebbe disfunzionale. Il processo di rimozione degli introni e di riconnessione degli esoni è chiamato splicing ( Figura sotto ). Gli introni vengono rimossi e degradati mentre il pre-mRNA è ancora nel nucleo. Lo splicing avviene tramite un meccanismo specifico della sequenza che assicura che gli introni vengano rimossi e gli esoni riuniti con l'accuratezza e la precisione di un singolo nucleotide. Sebbene l'introne stesso sia non codificante, l'inizio e la fine di ogni introne sono contrassegnati con nucleotidi specifici: GU all'estremità 5' e AG all'estremità 3' dell'introne. Lo splicing dei pre-mRNA è condotto da complessi di proteine e molecole di RNA chiamati spliceosomi.

Gli errori nello splicing sono implicati nei tumori e in altre malattie umane. Quali tipi di mutazioni potrebbero portare a errori nello splicing? Pensa a diversi possibili risultati se si verificano errori nello splicing.
Si noti che possono essere presenti più di 70 introni individuali e ognuno di essi deve subire il processo di splicing, oltre al capping 5' e all'aggiunta di una coda poli-A, solo per generare una singola molecola di mRNA traducibile.
-
introni ed esoni
-
introni ed esoni
-
struttura del cappuccio G
-
Struttura dell'mRNA
-
due nucleotidi con adenina
-
Schema dell'organizzazione delle giunzioni esone-introne-esone. Gli esoni a monte e a valle sono in arancione e l' introne è in grigio. Sono indicati i nucleotidi conservati.
-
Meccanismo di eliminazione di un introne
Elaborazione di tRNA e rRNA
[modifica | modifica sorgente]I tRNA e gli rRNA sono molecole strutturali che hanno un ruolo nella sintesi proteica; tuttavia, questi RNA non vengono tradotti di per sé. I pre-rRNA vengono trascritti, elaborati e assemblati in ribosomi nel nucleolo. I pre-tRNA vengono trascritti ed elaborati nel nucleo e poi rilasciati nel citoplasma dove vengono legati ad amminoacidi liberi per la sintesi proteica.
La maggior parte dei tRNA e degli rRNA negli eucarioti e nei procarioti vengono prima trascritti come una lunga molecola precursore che si estende su più rRNA o tRNA. Gli enzimi scindono quindi i precursori in subunità corrispondenti a ciascun RNA strutturale. Alcune delle basi dei pre-rRNA sono metilate ; ovvero, viene aggiunto un gruppo funzionale metilico –CH 3 per la stabilità. Anche le molecole di pre-tRNA subiscono metilazione. Come per i pre-mRNA, l'escissione delle subunità avviene nei pre-RNA eucariotici destinati a diventare tRNA o rRNA.
Gli rRNA maturi costituiscono circa il 50 percento di ogni ribosoma. Alcune delle molecole di RNA di un ribosoma sono puramente strutturali, mentre altre hanno attività catalitiche o di legame. I tRNA maturi assumono una struttura tridimensionale attraverso regioni locali di appaiamento delle basi stabilizzate dal legame idrogeno intramolecolare. Il tRNA si ripiega per posizionare il sito di legame dell'amminoacido a un'estremità e l' anticodone all'altra estremità ( Figura sotto). L'anticodone è una sequenza di tre nucleotidi in un tRNA che interagisce con un codone di mRNA attraverso l'appaiamento complementare delle basi.

.
I ribosomi e la sintesi proteica
[modifica | modifica sorgente]La sintesi delle proteine consuma più energia di una cellula rispetto a qualsiasi altro processo metabolico. A loro volta, le proteine rappresentano più massa di qualsiasi altro componente degli organismi viventi (ad eccezione dell'acqua) e svolgono praticamente ogni funzione di una cellula. Il processo di traduzione, o sintesi proteica, comporta la decodifica di un messaggio di mRNA in un prodotto polipeptidico. Gli amminoacidi sono legati insieme in modo covalente mediante legami peptidici interconnessi in lunghezze che vanno da circa 50 a più di 1000 residui di amminoacidi. Ogni singolo amminoacido ha un gruppo amminico (NH 2 ) e un gruppo carbossilico (COOH). I polipeptidi si formano quando il gruppo amminico di un amminoacido forma un legame ammidico (cioè peptidico) con il gruppo carbossilico di un altro amminoacido ( Figura 15.15 ). Questa reazione è catalizzata dai ribosomi e genera una molecola d'acqua.

Il meccanismo di sintesi proteica
[modifica | modifica sorgente]Oltre al modello di mRNA, molte molecole e macromolecole contribuiscono al processo di traduzione. La composizione di ogni componente può variare tra le specie; ad esempio, i ribosomi possono essere costituiti da un numero diverso di rRNA e polipeptidi a seconda dell'organismo. Tuttavia, le strutture e le funzioni generali del macchinario di sintesi proteica sono paragonabili dai batteri alle cellule umane. La traduzione richiede l'input di un modello di mRNA, ribosomi, tRNA e vari fattori enzimatici. (Nota: un ribosoma può essere pensato come un enzima i cui siti di legame degli amminoacidi sono specificati dall'mRNA.)
Ribosomi
[modifica | modifica sorgente]
Anche prima che un mRNA venga tradotto, una cellula deve investire energia per costruire ciascuno dei suoi ribosomi. In E. coli , ci sono tra 10.000 e 70.000 ribosomi presenti in ogni cellula in un dato momento. Un ribosoma è una macromolecola complessa composta da rRNA strutturali e catalitici e molti polipeptidi distinti. Negli eucarioti, il nucleolo è completamente specializzato per la sintesi e l'assemblaggio di rRNA.
I ribosomi sono presenti nel citoplasma dei procarioti e nel citoplasma e nel reticolo endoplasmatico rugoso degli eucarioti. Anche i mitocondri e i cloroplasti hanno i loro ribosomi nella matrice e nello stroma, che sono più simili ai ribosomi procariotici (e hanno simili sensibilità ai farmaci) rispetto ai ribosomi appena fuori dalle loro membrane esterne nel citoplasma. I ribosomi si dissociano in subunità grandi e piccole quando non sintetizzano proteine e si riassociano durante l'inizio della traduzione. In E. coli, la subunità piccola è descritta come 30S e la subunità grande è 50S, per un totale di 70S (ricorda che le unità di Svedberg non sono additive). I ribosomi dei mammiferi hanno una subunità piccola 40S e una subunità grande 60S, per un totale di 80S. La subunità piccola è responsabile del legame con lo stampo dell'mRNA, mentre la subunità grande lega sequenzialmente i tRNA. Ogni molecola di mRNA viene tradotta simultaneamente da molti ribosomi, tutti sintetizzanti la proteina nella stessa direzione: leggendo l'mRNA da 5' a 3' e sintetizzando il polipeptide dal terminale N al terminale C. La struttura completa mRNA/poliribosoma è chiamata polisoma .
tRNA
[modifica | modifica sorgente]I tRNA sono molecole di RNA strutturali che sono state trascritte dai geni dalla RNA polimerasi III. A seconda della specie, nel citoplasma esistono da 40 a 60 tipi di tRNA. Gli RNA di trasferimento servono come molecole adattatrici. Ogni tRNA trasporta uno specifico amminoacido e riconosce uno o più codoni di mRNA che definiscono l'ordine degli amminoacidi in una proteina. Gli amminoacil-tRNA si legano al ribosoma e aggiungono l'amminoacido corrispondente alla catena polipeptidica. Pertanto, i tRNA sono le molecole che effettivamente "traducono" il linguaggio dell'RNA nel linguaggio delle proteine.
-
tRNA: struttura secondaria
-
tRNA: struttura terziaria
-
tRNA: struttura
-
tRNA 3d
-
tRNA: modello atomico
-
tRNA complessato con la sua aminoacil-tRNA sintetasi
Dei 64 possibili codoni mRNA, o combinazioni di triplette di A, U, G e C, tre specificano la terminazione della sintesi proteica e 61 specificano l'aggiunta di amminoacidi alla catena polipeptidica. Di questi 61, un codone (AUG) codifica anche l'inizio della traduzione. Ogni anticodone tRNA può formare una coppia di basi con uno o più codoni mRNA per il suo amminoacido. Ad esempio, se la sequenza CUA si verificasse su un modello mRNA nel frame di lettura appropriato, si legherebbe a un tRNA leucina che esprime la sequenza complementare, GAU. La capacità di alcuni tRNA di corrispondere a più di un codone è ciò che conferisce al codice genetico la sua struttura a blocchi.
Come molecole adattatrici della traduzione, è sorprendente che i tRNA riescano a contenere così tanta specificità in un pacchetto così piccolo. Considerate che i tRNA devono interagire con tre fattori: 1) devono essere riconosciuti dalla corretta aminoacil sintetasi (vedere sotto); 2) devono essere riconosciuti dai ribosomi; e 3) devono legarsi alla sequenza corretta nell'mRNA.

Aminoacil tRNA sintetasi
[modifica | modifica sorgente]Il processo di sintesi del pre-tRNA da parte della RNA polimerasi III crea solo la porzione di RNA della molecola adattatrice. L'amminoacido corrispondente deve essere aggiunto in seguito, una volta che il tRNA è stato elaborato ed esportato nel citoplasma. Attraverso il processo di "carica" del tRNA, ogni molecola di tRNA è legata al suo amminoacido corretto da uno di un gruppo di enzimi chiamati amminoacil tRNA sintetasi . Esiste almeno un tipo di amminoacil tRNA sintetasi per ciascuno dei 20 amminoacidi; il numero esatto di amminoacil tRNA sintetasi varia a seconda della specie. Questi enzimi prima legano e idrolizzano l'ATP per catalizzare un legame ad alta energia tra un amminoacido e l'adenosina monofosfato (AMP); una molecola di pirofosfato viene espulsa in questa reazione. L'amminoacido attivato viene quindi trasferito al tRNA e viene rilasciato l'AMP. Il termine "carica" è appropriato, poiché il legame ad alta energia che collega un amminoacido al suo tRNA viene in seguito utilizzato per guidare la formazione del legame peptidico. Ogni tRNA prende il nome dal suo amminoacido.

Il meccanismo della sintesi proteica
[modifica | modifica sorgente]Come per la sintesi di mRNA, la sintesi proteica può essere divisa in tre fasi: inizio, allungamento e terminazione . Il processo di traduzione è simile nei procarioti e negli eucarioti. Qui esploreremo come avviene la traduzione in E. coli , un procariote rappresentativo, e specificheremo eventuali differenze tra la traduzione procariotica ed eucariotica.
Inizio della traduzione
[modifica | modifica sorgente]La sintesi proteica inizia con la formazione di un complesso di inizio . In E. coli , questo complesso coinvolge il piccolo ribosoma 30S, il modello di mRNA, tre fattori di inizio (IF; IF-1, IF-2 e IF-3) e uno speciale tRNA iniziatore , chiamato tRNA fMet .
Nell'mRNA di E. coli , una sequenza a monte del primo codone AUG, chiamata sequenza Shine-Dalgarno (AGGAGG), interagisce con le molecole di rRNA che compongono il ribosoma. Questa interazione ancora la subunità ribosomiale 30S nella posizione corretta sullo stampo dell'mRNA. Il guanosina trifosfato (GTP), che è un nucleotide trifosfato purinico, agisce come fonte di energia durante la traduzione, sia all'inizio dell'allungamento che durante la traslocazione del ribosoma. Il legame dell'mRNA al ribosoma 30S richiede anche IF-3.
Il tRNA iniziatore interagisce quindi con il codone di inizio AUG (o raramente, GUG). Questo tRNA trasporta l'amminoacido metionina, che viene formilato dopo il suo legame al tRNA. La formilazione crea un legame peptidico "finto" tra il gruppo formil carbossilico e il gruppo amminico della metionina. Il legame del fMet-tRNA fMet è mediato dal fattore di inizio IF-2. Il fMet inizia ogni catena polipeptidica sintetizzata da E. coli , ma di solito viene rimosso dopo il completamento della traduzione. Quando un AUG in-frame viene incontrato durante l'allungamento della traduzione, una metionina non formilata viene inserita da un Met-tRNA Met regolare . Dopo la formazione del complesso di inizio, la subunità ribosomiale 30S viene unita alla subunità 50S per formare il complesso di traduzione. Negli eucarioti, si forma un complesso di inizio simile, comprendente mRNA, la piccola subunità ribosomiale 40S, IF eucariotici e nucleosidi trifosfati (GTP e ATP). La metionina sul tRNA iniziatore carico, chiamato Met-tRNA i , non è formilata. Tuttavia, Met-tRNA i è distinto da altri Met-tRNA in quanto può legare IF.
Invece di depositarsi nella sequenza Shine-Dalgarno, il complesso di inizio eucariotico riconosce il cappuccio di 7-metilguanosina all'estremità 5' dell'mRNA. Una proteina legante il cappuccio (CBP) e diversi altri IF assistono il movimento del ribosoma verso il cappuccio 5'. Una volta al cappuccio, il complesso di inizio segue l'mRNA nella direzione 5'-3', alla ricerca del codone di inizio AUG. Molti mRNA eucariotici vengono tradotti dal primo AUG, ma non è sempre così. Secondo le regole di Kozak , i nucleotidi attorno all'AUG indicano se si tratta del codone di inizio corretto. Le regole di Kozak stabiliscono che la seguente sequenza di consenso deve apparire attorno all'AUG dei geni dei vertebrati: 5'-gccRccAUGG-3'. La R (per purina) indica un sito che può essere A o G, ma non può essere C o U. In sostanza, più la sequenza è vicina a questo consenso, maggiore è l'efficienza della traduzione.
Una volta identificato l'AUG appropriato, le altre proteine e la CBP si dissociano e la subunità 60S si lega al complesso di Met-tRNA i , mRNA e subunità 40S. Questo passaggio completa l'inizio della traduzione negli eucarioti.
Traduzione, allungamento e terminazione
[modifica | modifica sorgente]Nei procarioti e negli eucarioti, le basi dell'allungamento sono le stesse, quindi esamineremo l'allungamento dal punto di vista di E. coli . Quando si forma il complesso di traduzione, la regione di legame del tRNA del ribosoma è composta da tre compartimenti. Il sito A (amminoacilico) lega i tRNA amminoacilici carichi in arrivo. Il sito P (peptidilico) lega i tRNA carichi che trasportano amminoacidi che hanno formato legami peptidici con la catena polipeptidica in crescita ma non si sono ancora dissociati dal loro tRNA corrispondente. Il sito E (di uscita) rilascia i tRNA dissociati in modo che possano essere ricaricati con amminoacidi liberi. Il metionil-tRNA di avvio, tuttavia, occupa il sito P all'inizio della fase di allungamento della traduzione sia nei procarioti che negli eucarioti.
Durante l'allungamento della traduzione, il modello di mRNA fornisce specificità di legame al tRNA. Mentre il ribosoma si muove lungo l'mRNA, ogni codone di mRNA entra in registro e viene assicurato il legame specifico con il corrispondente anticodone di tRNA carico. Se l'mRNA non fosse presente nel complesso di allungamento, il ribosoma legherebbe i tRNA in modo non specifico e casuale.
L'allungamento procede con tRNA carichi che entrano ed escono sequenzialmente dal ribosoma man mano che ogni nuovo amminoacido viene aggiunto alla catena polipeptidica. Il movimento di un tRNA dal sito A al sito P al sito E è indotto da cambiamenti conformazionali che fanno avanzare il ribosoma di tre basi nella direzione 3'. L'energia per ogni passaggio lungo il ribosoma è donata da fattori di allungamento che idrolizzano il GTP. L'energia del GTP è richiesta sia per il legame di un nuovo amminoacil-tRNA al sito A sia per la sua traslocazione al sito P dopo la formazione del legame peptidico. I legami peptidici si formano tra il gruppo amminico dell'amminoacido attaccato al tRNA del sito A e il gruppo carbossilico dell'amminoacido attaccato al tRNA del sito P. La formazione di ogni legame peptidico è catalizzata dalla peptidil transferasi , un enzima basato sull'RNA che è integrato nella subunità ribosomiale 50S. L'energia per ogni formazione di legame peptidico deriva dal legame ad alta energia che collega ogni amminoacido al suo tRNA. Dopo la formazione del legame peptidico, il tRNA del sito A che ora contiene la catena peptidica in crescita si sposta nel sito P, e il tRNA del sito P che ora è vuoto si sposta nel sito E e viene espulso dal ribosoma ( Figura sotto). Incredibilmente, l' apparato di traduzione di E. coli impiega solo 0,05 secondi per aggiungere ogni amminoacido, il che significa che una proteina di 200 amminoacidi può essere tradotta in soli 10 secondi.

| Esercizio |
|---|
| Molti antibiotici inibiscono la sintesi proteica batterica. Ad esempio, la tetraciclina blocca il sito A sul ribosoma batterico e il cloramfenicolo blocca il trasferimento peptidilico. Quale effetto specifico ti aspetteresti che ciascuno di questi antibiotici avesse sulla sintesi proteica?
La tetraciclina influenzerebbe direttamente:
Il cloramfenicolo influenzerebbe direttamente:
|
La terminazione della traduzione avviene quando si incontra un codone nonsenso (UAA, UAG o UGA). Dopo l'allineamento con il sito A, questi codoni nonsenso vengono riconosciuti da fattori di rilascio proteico che assomigliano ai tRNA. I fattori di rilascio sia nei procarioti che negli eucarioti istruiscono la peptidil transferasi ad aggiungere una molecola d'acqua all'estremità carbossilica dell'amminoacido del sito P. Questa reazione costringe l'amminoacido del sito P a staccarsi dal suo tRNA e la proteina appena creata viene rilasciata. Le subunità ribosomiali piccole e grandi si dissociano dall'mRNA e l'una dall'altra; vengono reclutate quasi immediatamente in un altro complesso di inizio della traduzione. Dopo che molti ribosomi hanno completato la traduzione, l'mRNA viene degradato in modo che i nucleotidi possano essere riutilizzati in un'altra reazione di trascrizione.

-
schema generale della traduzione (sintesi delle proteine)
-
sintesi della proteina
Piegatura, modifica e indirizzamento delle proteine
[modifica | modifica sorgente]Durante e dopo la traduzione, singoli amminoacidi possono essere modificati chimicamente, sequenze segnale aggiunte e la nuova proteina "piegata" in una distinta struttura tridimensionale come risultato di interazioni intramolecolari. Una sequenza segnale è una breve sequenza all'estremità amminica di una proteina che la indirizza a uno specifico compartimento cellulare. Queste sequenze possono essere considerate il "biglietto del treno" della proteina per la sua destinazione finale e sono riconosciute da proteine di riconoscimento del segnale che agiscono come conduttori. Ad esempio, un termine specifico della sequenza segnale indirizzerà una proteina ai mitocondri o ai cloroplasti (nelle piante). Una volta che la proteina raggiunge la sua destinazione cellulare, la sequenza segnale viene solitamente tagliata.
Molte proteine si ripiegano spontaneamente, ma alcune proteine necessitano di molecole di supporto, chiamate chaperoni , per impedire loro di aggregarsi durante il complicato processo di ripiegamento. Anche se una proteina è correttamente specificata dal suo mRNA corrispondente, potrebbe assumere una forma completamente disfunzionale se condizioni anomale di temperatura o pH impediscono il corretto ripiegamento.

| Funzione peptidica | Composizione |
|---|---|
| Trasporto al nucleo cellulare ( NLS ) | -Pro-Pro-Light-Light-Light-Arg-Light-Val- |
| Trasporto al reticolo endoplasmatico | H 2 N-Met-Met-Ser-Phe-Val-Ser-Leu-Leu-Leu-Val-Gly-Ile-Leu-Phe-Trp-Ala-Thr-Glu-Ala-Glu-Gln-Leu-Thr- Lys-Cys-Glu-Val-Phe-Gln- |
| Ritenzione nel reticolo endoplasmatico | -Lys-Asp-Glu-Leu-COOH |
| Trasporto alla matrice mitocondriale | H 2 N-Met-Leu-Ser-Leu-Arg-Gln-Ser-Ile-Arg-Phe-Phe-Lys-Pro-Ala-Thr-Arg-Thr-Leu-Cys-Ser-Ser-Arg-Tyr- Leu-Leu- |
| Trasporto al perossisoma (PTS1) | -Ser-Lys-Leu-COOH |
| Trasporto al perossisoma (PTS2) | H 2 N-----Arg-Leu-X 5 -His-Leu- |
H2N è il terminale N di una proteina e COOH il suo terminale C.
-
clivaggio (taglio) dell'insulina
-
glicosilazione di una proteina
-
Modifiche post-traduzionali alle proteine
-
Il proteasoma degrada le proteine
-
Proteasoma visto dall'alto