Biologia per il liceo/La genetica post-mendeliana
Alternative alla dominanza e alla recessività
[modifica | modifica sorgente]Gli esperimenti di Mendel con le piante di pisello suggerivano che: (1) esistono due "unità" o alleli per ogni gene; (2) gli alleli mantengono la loro integrità in ogni generazione (nessuna fusione); e (3) in presenza dell'allele dominante, l'allele recessivo è nascosto e non contribuisce al fenotipo. Pertanto, gli alleli recessivi possono essere "portati" e non espressi dagli individui. Tali individui eterozigoti sono talvolta indicati come "portatori". Ulteriori studi genetici su altre piante e animali hanno dimostrato che esiste una complessità molto maggiore, ma che i principi fondamentali della genetica mendeliana sono ancora validi. Nelle sezioni che seguono, considereremo alcune delle estensioni del mendelismo. Se Mendel avesse scelto un sistema sperimentale che esibisse queste complessità genetiche, è possibile che non avrebbe capito cosa significassero i suoi risultati.
Dominanza incompleta
[modifica | modifica sorgente]I risultati di Mendel, secondo cui i tratti sono ereditati come coppie dominanti e recessive, contraddicevano la visione di quel tempo secondo cui la prole esibiva una miscela dei tratti dei genitori. Tuttavia, il fenotipo eterozigote occasionalmente sembra essere intermedio tra i due genitori. Ad esempio, nello snapdragon, Antirrhinum majus ( Figura sotto), un incrocio tra un genitore omozigote con fiori bianchi (CWCW) e un genitore omozigote con fiori rossi (CRCR) produrrà una prole con fiori rosa (CRCW). (Si noti che vengono utilizzate diverse abbreviazioni genotipiche per le estensioni mendeliane per distinguere questi modelli dalla semplice dominanza e recessività.) Questo modello di ereditarietà è descritto come dominanza incompleta , che denota l'espressione di due alleli contrastanti tali che l'individuo mostra un fenotipo intermedio. L'allele per i fiori rossi è incompletamente dominante sull'allele per i fiori bianchi. Tuttavia, i risultati di un autoincrocio eterozigote possono ancora essere previsti, proprio come con gli incroci dominanti e recessivi mendeliani. In questo caso, il rapporto genotipico sarebbe 1 CRCR : 2 CRCW : 1 CWCW e il rapporto fenotipico sarebbe 1:2:1 per rosso:rosa:bianco.
Codominanza
[modifica | modifica sorgente]Una variante della dominanza incompleta è la codominanza , in cui entrambi gli alleli per la stessa caratteristica sono espressi simultaneamente nell'eterozigote. Un esempio di codominanza sono i gruppi sanguigni MN degli esseri umani. Gli alleli M e N sono espressi sotto forma di un antigene M o N presente sulla superficie dei globuli rossi. Gli omozigoti ( LMLM e LNLN ) esprimono l'allele M o N, e gli eterozigoti ( LMLN ) esprimono entrambi gli alleli in egual modo. In un autoincrocio tra eterozigoti che esprimono un tratto codominante, i tre possibili genotipi della prole sono fenotipicamente distinti. Tuttavia, il rapporto genotipico 1:2:1 caratteristico di un incrocio monoibrido mendeliano è ancora valido.

Alleli multipli
[modifica | modifica sorgente]Mendel ha ipotizzato che solo due alleli, uno dominante e uno recessivo, potessero esistere per un dato gene. Ora sappiamo che questa è una semplificazione eccessiva. Sebbene gli esseri umani individuali (e tutti gli organismi diploidi) possano avere solo due alleli per un dato gene, possono esistere più alleli a livello di popolazione, in modo tale che si osservino molte combinazioni di due alleli. Si noti che quando esistono molti alleli per lo stesso gene, la convenzione è quella di indicare il fenotipo o genotipo più comune tra gli animali selvatici come tipo selvatico (spesso abbreviato "+"); questo è considerato lo standard o la norma. Tutti gli altri fenotipi o genotipi sono considerati varianti di questo standard, il che significa che si discostano dal tipo selvatico. La variante può essere recessiva o dominante rispetto all'allele di tipo selvatico.
Un esempio di alleli multipli è il colore del mantello nei conigli ( Figura sotto). Qui, esistono quattro alleli per il gene c . La versione di tipo selvatico, C+C+ , è espressa come pelliccia scura. Il fenotipo del cincillà, cchcch , è espresso come pelliccia grigia. Il fenotipo himalayano, chch , ha pelliccia nera sulle estremità e pelliccia bianca altrove. Infine, il fenotipo albino, o "incolore", cc , è espresso come pelliccia bianca. In caso di alleli multipli, possono esistere gerarchie di dominanza. In questo caso, l'allele di tipo selvatico è dominante su tutti gli altri, il cincillà è incompletamente dominante su himalayano e albino, e l'himalayano è dominante su albino. Questa gerarchia, o serie allelica, è stata rivelata osservando i fenotipi di ogni possibile prole eterozigote.

Figura sopra. La completa dominanza di un fenotipo di tipo selvatico su tutti gli altri mutanti spesso si verifica come effetto del "dosaggio" di uno specifico prodotto genico, in modo tale che l'allele di tipo selvatico fornisca la giusta quantità di prodotto genico mentre gli alleli mutanti non possono. Per la serie allelica nei conigli, l'allele di tipo selvatico può fornire un dato dosaggio di pigmento della pelliccia, mentre i mutanti forniscono un dosaggio inferiore o nessuno. È interessante notare che il fenotipo himalayano è il risultato di un allele che produce un prodotto genico sensibile alla temperatura che produce pigmento solo nelle estremità più fredde del corpo del coniglio.
In alternativa, un allele mutante può essere dominante su tutti gli altri fenotipi, incluso il tipo selvatico. Ciò può verificarsi quando l'allele mutante interferisce in qualche modo con il messaggio genetico, in modo che anche un eterozigote con una copia dell'allele di tipo selvatico esprima il fenotipo mutante. Un modo in cui l'allele mutante può interferire è potenziando la funzione del prodotto genico di tipo selvatico o modificandone la distribuzione nel corpo. Un esempio di ciò è la mutazione Antennapedia in Drosophila ( Figura sotto). In questo caso, l'allele mutante espande la distribuzione del prodotto genico e, di conseguenza, l' eterozigote Antennapedia sviluppa zampe sulla testa dove dovrebbero esserci le sue antenne.

Caratteri legati al cromosoma X
[modifica | modifica sorgente]Negli esseri umani, così come in molti altri animali e in alcune piante, il sesso dell'individuo è determinato dai cromosomi sessuali. I cromosomi sessuali sono una coppia di cromosomi non omologhi. Finora, abbiamo preso in considerazione solo i modelli di ereditarietà tra cromosomi non sessuali, o autosomi . Oltre a 22 coppie omologhe di autosomi, le femmine umane hanno una coppia omologa di cromosomi X, mentre i maschi umani hanno una coppia di cromosomi XY. Sebbene il cromosoma Y contenga una piccola regione di somiglianza con il cromosoma X in modo che possano accoppiarsi durante la meiosi, il cromosoma Y è molto più corto e contiene molti meno geni. Infatti, quando Nettie Stevens scoprì che i cromosomi X e Y erano i determinanti del sesso, li differenziò solo in base alle dimensioni. (Si noti che in questo caso e nella descrizione sottostante, i termini cromosoma X e cromosoma Y non erano utilizzati all'epoca.) Quando un gene esaminato è presente sul cromosoma X, ma non su quello Y, si dice che è legato al cromosoma X.
Il colore degli occhi in Drosophila è stato uno dei primi tratti legati al cromosoma X ad essere identificato. Thomas Hunt Morgan ha mappato questo tratto su quello che è diventato noto come cromosoma X nel 1910. Come gli esseri umani, i maschi di Drosophila hanno una coppia di cromosomi XY e le femmine sono XX. Nei moscerini, il colore degli occhi di tipo selvatico è rosso (XW ) ed è dominante sul colore degli occhi bianco (Xw ) ( Figura sotto ). A causa della posizione del gene del colore degli occhi, gli incroci reciproci non producono gli stessi rapporti di prole. I maschi sono detti emizigoti , perché hanno un solo allele per ogni caratteristica legata al cromosoma X. L'emizigosità rende le descrizioni di dominanza e recessività irrilevanti per i maschi XY. I maschi di Drosophila non hanno una seconda copia di allele sul cromosoma Y; cioè, il loro genotipo può essere solo XWY o XwY. Al contrario, le femmine hanno due copie alleliche di questo gene e possono essere XW XW , XWXw , o Xw Xw .

In un incrocio legato al cromosoma X, i genotipi della prole F1 e F2 dipendono dal fatto che il tratto recessivo sia stato espresso dal maschio o dalla femmina nella generazione P1. Per quanto riguarda il colore degli occhi di Drosophila , quando il maschio P1 esprime il fenotipo occhi bianchi e la femmina è omozigote occhi rossi, tutti i membri della generazione F1 presentano occhi rossi ( Figura 12.12 ). Le femmine F1 sono eterozigote (XWXw ), e i maschi sono tutti XWY, avendo ricevuto il loro cromosoma X dalla femmina omozigote dominante P1 e il loro cromosoma Y dal maschio P1. Un successivo incrocio tra la femmina XWXw e il maschio XWY produrrebbe solo femmine occhi rossi (con genotipi XWXW o XWXw ) e maschi sia occhi rossi che bianchi (con genotipi XWY o XwY ). Ora, considera un incrocio tra una femmina omozigote con occhi bianchi e un maschio con occhi rossi. La generazione F1 presenterebbe solo femmine eterozigote con occhi rossi (XWXw) e solo maschi con occhi bianchi (X w Y). Metà delle femmine F2 avrebbe occhi rossi (XWXw) e metà avrebbe occhi bianchi (XWXW ). Allo stesso modo, metà dei maschi F2 avrebbe occhi rossi (XWY) e metà avrebbe occhi bianchi (XwY).

Le scoperte nella genetica del moscerino della frutta possono essere applicate alla genetica umana. Quando una madre è omozigote per un tratto recessivo legato al cromosoma X, trasmetterà il tratto al 100 percento della sua prole. La sua prole maschile è, quindi, destinata a esprimere il tratto, poiché erediteranno il cromosoma Y del padre. Negli esseri umani, gli alleli per alcune condizioni (alcune forme di daltonismo, emofilia e distrofia muscolare) sono legati al cromosoma X. Le femmine eterozigote per queste malattie sono considerate portatrici e potrebbero non presentare alcun effetto fenotipico. Queste femmine trasmetteranno la malattia a metà dei loro figli maschi e trasmetteranno lo stato di portatrici a metà delle loro figlie; pertanto, i tratti recessivi legati al cromosoma X compaiono più frequentemente nei maschi che nelle femmine.
In alcuni gruppi di organismi con cromosomi sessuali, il sesso con i cromosomi sessuali non omologhi è la femmina piuttosto che il maschio. Questo è il caso di tutti gli uccelli. In questo caso, i tratti legati al sesso avranno maggiori probabilità di apparire nella femmina, in cui sono emizigoti.
Disturbi umani legati al sesso
[modifica | modifica sorgente]Gli studi di collegamento sessuale nel laboratorio di Morgan hanno fornito le basi per comprendere i disturbi recessivi legati al cromosoma X negli esseri umani, che includono il daltonismo rosso-verde e l'emofilia di tipo A e B. Poiché i maschi umani devono ereditare solo un allele mutante recessivo X per essere colpiti, i disturbi legati al cromosoma X sono osservati in modo sproporzionato nei maschi. Le femmine devono ereditare alleli recessivi legati al cromosoma X da entrambi i genitori per esprimere il tratto. Quando ereditano un allele mutante recessivo legato al cromosoma X e un allele selvaggio dominante legato al cromosoma X, sono portatrici del tratto e in genere non sono colpite. Le femmine portatrici possono manifestare forme lievi del tratto a causa dell'inattivazione dell'allele dominante situato su uno dei cromosomi X. Tuttavia, le portatrici femmine possono trasmettere il tratto ai loro figli maschi, con il risultato che il maschio esibisce il tratto, oppure possono trasmettere l'allele recessivo alle loro figlie femmine, con il risultato che i figli sono portatori del tratto ( Figura sotto ). Sebbene esistano alcuni disturbi recessivi legati all'Y, in genere sono associati all'infertilità nei maschi e pertanto non vengono trasmessi alle generazioni successive.

.
I geni collegati violano la legge dell'assortimento indipendente
[modifica | modifica sorgente]Sebbene tutte le caratteristiche dei piselli di Mendel si comportassero secondo la legge dell'assortimento indipendente, ora sappiamo che alcune combinazioni di alleli non vengono ereditate indipendentemente l'una dall'altra. I geni che si trovano su cromosomi non omologhi separati si ordineranno sempre in modo indipendente. Tuttavia, ogni cromosoma contiene centinaia o migliaia di geni, organizzati linearmente sui cromosomi come perle su una collana. La segregazione degli alleli nei gameti può essere influenzata dal collegamento , in cui i geni che si trovano fisicamente vicini l'uno all'altro sullo stesso cromosoma hanno maggiori probabilità di essere ereditati in coppia. Tuttavia, a causa del processo di ricombinazione, o "crossover", è possibile che due geni sullo stesso cromosoma si comportino in modo indipendente, o come se non fossero collegati. Per capirlo, consideriamo le basi biologiche del collegamento e della ricombinazione genica.
I cromosomi omologhi possiedono gli stessi geni nello stesso ordine lineare. Gli alleli possono differire sulle coppie di cromosomi omologhi, ma i geni a cui corrispondono non lo fanno. In preparazione alla prima divisione della meiosi, i cromosomi omologhi si replicano e formano sinapsi. Geni simili sugli omologhi si allineano tra loro. In questa fase, segmenti di cromosomi omologhi scambiano segmenti lineari di materiale genetico ( Figura sotto). Questo processo è chiamato ricombinazione o crossing-over, ed è un processo genetico comune. Poiché i geni sono allineati durante la ricombinazione, l'ordine dei geni non viene alterato. Invece, il risultato della ricombinazione è che gli alleli materni e paterni vengono combinati sullo stesso cromosoma. In un dato cromosoma, possono verificarsi diversi eventi di ricombinazione, causando un ampio rimescolamento degli alleli.
-
Confronto, in un incrocio diibrido, di una F2 nel caso di geni collegati o non
-
Il processo di crossover, o ricombinazione, avviene quando due cromosomi omologhi si allineano durante la meiosi e scambiano un segmento di materiale genetico
Quando due geni si trovano in stretta prossimità sullo stesso cromosoma, sono considerati collegati e i loro alleli tendono a essere trasmessi insieme tramite meiosi. Per esemplificare questo, immagina un incrocio diibrido che coinvolge il colore del fiore e l'altezza della pianta in cui i geni sono uno accanto all'altro sul cromosoma. Se un cromosoma omologo ha alleli per piante alte e fiori rossi e l'altro cromosoma ha geni per piante basse e fiori gialli, allora quando si formano i gameti, gli alleli alti e rossi andranno insieme in un gamete e gli alleli bassi e gialli andranno in altri gameti. Questi sono chiamati genotipi parentali perché sono stati ereditati intatti dai genitori dell'individuo che produce gameti. Ma a differenza di quanto accadrebbe se i geni fossero su cromosomi diversi, non ci saranno gameti con alleli alti e gialli e nessun gamete con alleli bassi e rossi. Se crei il quadrato di Punnett con questi gameti, vedrai che la classica previsione mendeliana di un risultato 9:3:3:1 di un incrocio diibrido non si applicherebbe. Man mano che la distanza tra due geni aumenta, aumenta la probabilità di uno o più incroci tra di essi e i geni si comportano più come se fossero su cromosomi separati. I genetisti hanno utilizzato la proporzione di gameti ricombinanti (quelli non come i genitori) come misura di quanto siano distanti i geni su un cromosoma. Utilizzando queste informazioni, hanno costruito mappe elaborate di geni sui cromosomi per organismi ben studiati, compresi gli esseri umani.
La pubblicazione fondamentale di Mendel non fa menzione del linkage, e molti ricercatori hanno messo in dubbio che abbia incontrato il linkage ma abbia scelto di non pubblicare quegli incroci per timore che avrebbero invalidato il suo postulato di assortimento indipendente. Il pisello da giardino ha sette coppie di cromosomi, e alcuni hanno suggerito che la sua scelta di sette caratteristiche non fosse una coincidenza. Tuttavia, anche se i geni da lui esaminati non si trovassero su cromosomi separati, è possibile che semplicemente non abbia osservato il linkage a causa degli effetti di rimescolamento estesi della ricombinazione.
Epistasia
[modifica | modifica sorgente]Gli studi di Mendel sulle piante di pisello implicavano che la somma del fenotipo di un individuo fosse controllata dai geni (o come li chiamava lui, fattori unitari), in modo tale che ogni caratteristica fosse distintamente e completamente controllata da un singolo gene. Infatti, le singole caratteristiche osservabili sono quasi sempre sotto l'influenza di più geni (ciascuno con due o più alleli) che agiscono all'unisono. Ad esempio, almeno otto geni contribuiscono al colore degli occhi negli esseri umani.
In alcuni casi, diversi geni possono contribuire ad aspetti di un fenotipo comune senza che i loro prodotti genici interagiscano mai direttamente. Nel caso dello sviluppo di un organo, ad esempio, i geni possono essere espressi in sequenza, con ogni gene che aggiunge complessità e specificità all'organo. I geni possono funzionare in modo complementare o sinergico, in modo tale che due o più geni debbano essere espressi simultaneamente per influenzare un fenotipo. I geni possono anche opporsi l'uno all'altro, con un gene che modifica l'espressione di un altro.
Nell'epistasi , l'interazione tra geni è antagonista, in modo tale che un gene maschera o interferisce con l'espressione di un altro. " Epistasi " è una parola composta da radici greche che significano "stare sopra". Gli alleli che vengono mascherati o silenziati sono detti ipostatici rispetto agli alleli epistatici che stanno eseguendo il mascheramento. Spesso la base biochimica dell'epistasi è un percorso genico in cui l'espressione di un gene dipende dalla funzione di un gene che lo precede o lo segue nel percorso.
Un esempio di epistasi è la pigmentazione nei topi. Il colore del mantello di tipo selvatico, agouti ( AA ), è dominante rispetto alla pelliccia tinta unita ( aa ). Tuttavia, un gene separato ( C ) è necessario per la produzione di pigmento. Un topo con un allele recessivo c in questo locus non è in grado di produrre pigmento ed è albino indipendentemente dall'allele presente nel locus A ( Figura sotto). Pertanto, i genotipi AAcc , Aacc e aacc producono tutti lo stesso fenotipo albino. Un incrocio tra eterozigoti per entrambi i geni ( AaCc x AaCc ) genererebbe una prole con un rapporto fenotipico di 9 agouti:3 colore tinta unita:4 albino ( Figura sotto). In questo caso, il gene C è epistatico rispetto al gene A.

L'epistasia può verificarsi anche quando un allele dominante maschera l'espressione di un gene separato. Il colore del frutto nella zucchina estiva è espresso in questo modo. L'espressione recessiva omozigote del gene W ( ww ) accoppiata all'espressione dominante omozigote o eterozigote del gene Y ( YY o Yy ) genera frutti gialli, mentre il genotipo wwyy produce frutti verdi. Tuttavia, se una copia dominante del gene W è presente nella forma omozigote o eterozigote, la zucchina estiva produrrà frutti bianchi indipendentemente dagli alleli Y. Un incrocio tra eterozigoti bianchi per entrambi i geni ( WwYy × WwYy ) produrrebbe una prole con un rapporto fenotipico di 12 bianco:3 giallo:1 verde.
Infine, l'epistasia può essere reciproca in modo tale che entrambi i geni, quando presenti nella forma dominante (o recessiva), esprimano lo stesso fenotipo. Nella pianta della borsa del pastore ( Capsella bursa-pastoris ), la caratteristica della forma del seme è controllata da due geni in una relazione epistatica dominante. Quando i geni A e B sono entrambi omozigoti recessivi ( aabb ), i semi sono ovoidali. Se è presente l'allele dominante per uno di questi geni, il risultato sono semi triangolari. Vale a dire, ogni possibile genotipo diverso da aabb produce semi triangolari e un incrocio tra eterozigoti per entrambi i geni ( AaBb x AaBb ) produrrebbe una prole con un rapporto fenotipico di 15 triangolari:1 ovoidale.
Mentre lavori sui problemi di genetica, tieni presente che ogni singola caratteristica che si traduce in un rapporto fenotipico che totalizza 16 è tipica di un'interazione a due geni. Ricorda il modello di ereditarietà fenotipica per l'incrocio diibrido di Mendel, che considerava due geni non interagenti: 9:3:3:1. Allo stesso modo, ci aspetteremmo che anche le coppie di geni interagenti esibissero rapporti espressi in 16 parti. Nota che stiamo assumendo che i geni interagenti non siano collegati; si stanno ancora assortendo indipendentemente nei gameti.
Il ruolo dei cromosomi nell'ereditarietà
[modifica | modifica sorgente]Il gene è l'unità fisica dell'ereditarietà e i geni sono disposti in un ordine lineare sui cromosomi. Il comportamento e l'interazione dei cromosomi durante la meiosi spiegano, a livello cellulare, i modelli di ereditarietà che osserviamo nelle popolazioni. I disturbi genetici che comportano alterazioni nel numero o nella struttura dei cromosomi possono avere effetti drammatici e possono impedire lo sviluppo di un ovulo fecondato.
Molto prima che gli scienziati visualizzassero i cromosomi al microscopio, il padre della genetica moderna, Gregor Mendel, iniziò a studiare l'ereditarietà nel 1843. Con tecniche microscopiche migliorate alla fine del 1800, i biologi cellulari potevano colorare e visualizzare le strutture subcellulari con coloranti e osservare le loro azioni durante la divisione cellulare e la meiosi. Con ogni divisione mitotica, i cromosomi si replicavano, si condensavano da una massa nucleare amorfa (nessuna forma costante) in corpi distinti a forma di X (coppie di cromatidi fratelli identici) e migravano verso poli cellulari separati.
Teoria cromosomica dell'ereditarietà
[modifica | modifica sorgente]La speculazione che i cromosomi potessero essere la chiave per comprendere l'ereditarietà ha portato diversi scienziati a esaminare le pubblicazioni di Mendel e a rivalutare il suo modello in termini di comportamento dei cromosomi durante la mitosi e la meiosi. Nel 1902, Theodor Boveri osservò che lo sviluppo embrionale corretto del riccio di mare non si verifica se non sono presenti i cromosomi. Nello stesso anno, Walter Sutton osservò la separazione dei cromosomi in cellule figlie durante la meiosi ( Figura 13.2 ). Insieme, queste osservazioni hanno portato alla teoria cromosomica dell'ereditarietà , che ha identificato i cromosomi come il materiale genetico responsabile dell'ereditarietà mendeliana.
-
a - Waler Sutton
-
b - Theodor Boveri
-
c - Eleanor Carothers
-
Thomas Morgan
In galleria: (a) Walter Sutton e (b) Theodor Boveri svilupparono la teoria cromosomica dell'ereditarietà, che afferma che i cromosomi trasportano l'unità dell'ereditarietà (geni). Eleanor Carothers (c), fu la prima a fornire prove fisiche a sostegno della teoria.
La teoria cromosomica dell'ereditarietà era coerente con le leggi di Mendel, supportate dalle seguenti osservazioni:
- Durante la meiosi, le coppie di cromosomi omologhi migrano come strutture discrete, indipendenti dalle altre coppie di cromosomi.
- L'ordinamento dei cromosomi da ciascuna coppia di omologhi ai pre-gameti sembra essere casuale.
- Ogni genitore sintetizza gameti che contengono solo metà del loro corredo cromosomico.
- Sebbene i gameti maschili e femminili (spermatozoi e ovuli) differiscano per dimensioni e morfologia, hanno lo stesso numero di cromosomi, il che suggerisce un uguale contributo genetico da parte di ciascun genitore.
- Durante la fecondazione, i cromosomi gametici si combinano per produrre una prole con lo stesso numero di cromosomi dei genitori.
Nonostante la mancanza di prove dirette che i cromosomi trasmettano tratti, la correlazione convincente tra il comportamento dei cromosomi durante la meiosi e le leggi astratte di Mendel ha portato gli scienziati a proporre la teoria cromosomica dell'ereditarietà. I critici hanno sottolineato che gli individui avevano molti più tratti segreganti indipendenti di quanti ne avessero i cromosomi. Circa dieci anni dopo che la teoria fu proposta, Eleanor Carothers fu la prima a scoprire prove fisiche a sostegno; osservò un assortimento cromosomico indipendente nelle cavallette. Quindi, dopo diversi anni di incroci con il moscerino della frutta, Drosophila melanogaster , Thomas Hunt Morgan ha fornito ulteriori prove sperimentali a sostegno della teoria cromosomica dell'ereditarietà.
Geni collegati e distanze
[modifica | modifica sorgente]Il lavoro di Mendel suggeriva che i tratti sono ereditati indipendentemente l'uno dall'altro. Morgan identificò una chiara corrispondenza tra la segregazione degli alleli e il cromosoma X, suggerendo che la segregazione casuale dei cromosomi fosse la base fisica del modello di Mendel. Ciò dimostrò anche che i geni collegati interrompono i risultati previsti da Mendel. Il fatto che ogni cromosoma possa trasportare molti geni collegati spiega come gli individui possano avere molti più tratti di quanti cromosomi abbiano. Tuttavia, i ricercatori nel laboratorio di Morgan suggerirono che gli alleli posizionati sullo stesso cromosoma non venivano sempre ereditati insieme. Durante la meiosi, i geni collegati in qualche modo si sono scollegati.
Ricombinazione omologa
[modifica | modifica sorgente]Nel 1909, Frans Janssen osservò i chiasmi, il punto in cui i cromatidi sono in contatto tra loro e possono scambiarsi segmenti, prima della prima divisione meiotica. Suggerì che gli alleli si scollegassero e che i cromosomi scambiassero fisicamente segmenti. Quando i cromosomi si condensavano e si accoppiavano con i loro omologhi, sembravano interagire in punti distinti. Janssen suggerì che questi punti corrispondessero a regioni in cui i segmenti cromosomici si scambiavano. Ora sappiamo che l'accoppiamento e l'interazione tra cromosomi omologhi, o sinapsi, fa più che organizzare semplicemente gli omologhi per la migrazione verso cellule figlie separate. Quando sinaptati, i cromosomi omologhi subiscono scambi fisici reciproci nei loro bracci nella ricombinazione omologa , o più semplicemente, "crossing over".
Per comprendere meglio il tipo di risultati sperimentali che i ricercatori stavano ottenendo in quel periodo, si consideri un individuo eterozigote che ha ereditato alleli materni dominanti per due geni sullo stesso cromosoma (come A e B ) e due alleli paterni recessivi per quegli stessi geni (come a e b ). Se i geni sono collegati, ci si aspetterebbe che questo individuo produca gameti che sono AB o ab con un rapporto 1:1. Se i geni non sono collegati, l'individuo dovrebbe produrre gameti AB , Ab , aB e ab con frequenze uguali, secondo il concetto mendeliano di assortimento indipendente. Poiché corrispondono a nuove combinazioni di alleli, i genotipi Ab e aB sono tipi non parentali che derivano dalla ricombinazione omologa durante la meiosi. I tipi parentali sono progenie che presentano la stessa combinazione allelica dei loro genitori. Morgan e i suoi colleghi, tuttavia, hanno scoperto che quando hanno incrociato questi individui eterozigoti con un genitore recessivo omozigote ( AaBb × aabb ), si sono verificati sia casi parentali che non parentali. Ad esempio, potrebbero essere recuperati 950 figli che erano o AaBb o aabb , ma ne sarebbero derivati anche 50 che erano o Aabb o aaBb . Questi risultati hanno suggerito che il linkage si è verificato più spesso, ma una significativa minoranza di figli era il prodotto della ricombinazione.
Uno degli esperimenti condotti nel laboratorio di Morgan, che prevedeva l'incrocio di mosche per due caratteristiche, il colore del corpo (grigio o nero) e la forma delle ali (normale e vestigiale), ha dimostrato gli eventi di ricombinazione che portano allo sviluppo di fenotipi non parentali ( Figura sotto).

Questo schema (figura sopra) mostra i modelli di ereditarietà genica non collegata e collegata. In (a), due geni sono localizzati su cromosomi diversi, quindi durante la meiosi si verifica un assortimento indipendente. La prole ha le stesse probabilità di essere del tipo parentale (ereditando la stessa combinazione di tratti dei genitori) o di un tipo non parentale (ereditando una combinazione di tratti diversa da quella dei genitori). In (b), due geni sono molto vicini tra loro sullo stesso cromosoma, quindi non si verifica alcun crossing over tra di loro. Pertanto, i geni vengono sempre ereditati insieme e tutta la prole è del tipo parentale. In (c), due geni sono molto distanti sul cromosoma, quindi il crossing over si verifica durante ogni evento meiotico. La frequenza di ricombinazione sarà la stessa come se i geni fossero su cromosomi separati. (d) La frequenza di ricombinazione effettiva della lunghezza delle ali e del colore del corpo del moscerino della frutta osservata da Thomas Morgan nel 1912 era del 17 percento. Una frequenza di crossover compresa tra 0 e 50 percento indica che i geni si trovano sullo stesso cromosoma e che talvolta si verifica il crossover.
Esercizio: In un test cross per due caratteristiche come quella qui descritta, la frequenza prevista della prole ricombinante può essere del 60 percento? Perché o perché no?
Mappe genetiche
[modifica | modifica sorgente]Janssen non aveva la tecnologia per dimostrare il crossing over, quindi rimase un'idea astratta a cui gli scienziati non credevano ampiamente. Gli scienziati pensavano che i chiasmi fossero una variazione della sinapsi e non riuscivano a capire come i cromosomi potessero rompersi e ricongiungersi. Tuttavia, i dati erano chiari sul fatto che il collegamento non sempre si verificava. Alla fine, ci vollero un giovane studente universitario e una "notte in bianco" per chiarire matematicamente il problema del collegamento e della ricombinazione.
Nel 1913, Alfred Sturtevant, uno studente del laboratorio di Morgan, raccolse i risultati dei ricercatori del laboratorio e li portò a casa una sera per rifletterci sopra. La mattina dopo, aveva creato la prima "mappa cromosomica", una rappresentazione lineare dell'ordine dei geni e della distanza relativa su un cromosoma ( Figura sotto).

| Esercizio |
|---|
Quale delle seguenti affermazioni è vera?
|
Come mostra la Figura sopra, utilizzando la frequenza di ricombinazione per predire la distanza genetica, possiamo dedurre l'ordine relativo dei geni sul cromosoma 2. I valori rappresentano le distanze della mappa in centimorgan (cM), che corrispondono alle frequenze di ricombinazione (in percentuale). Pertanto, i geni per il colore del corpo (gray) e le dimensioni delle ali erano distanti 67,0 − 48,5 = 18,5 cM, il che indica che gli alleli materni e paterni per questi geni si ricombinano nel 18,5 percento della prole, in media.
Per costruire una mappa cromosomica, Sturtevant ha ipotizzato che i geni fossero ordinati in serie su cromosomi filiformi. Ha anche ipotizzato che l'incidenza della ricombinazione tra due cromosomi omologhi potesse verificarsi con uguale probabilità in qualsiasi punto lungo la lunghezza del cromosoma. Operando in base a queste ipotesi, Sturtevant ha postulato che gli alleli che erano molto distanti su un cromosoma avevano maggiori probabilità di dissociarsi durante la meiosi semplicemente perché c'era una regione più ampia su cui poteva verificarsi la ricombinazione. Al contrario, gli alleli che erano vicini tra loro sul cromosoma avevano maggiori probabilità di essere ereditati insieme. Il numero medio di incroci tra due alleli, ovvero la loro frequenza di ricombinazione , era correlato alla loro distanza genetica l'uno dall'altro, rispetto alle posizioni di altri geni su quel cromosoma. Considerando l'esempio di incrocio tra AaBb e aabb sopra, potremmo calcolare la frequenza di ricombinazione come 50/1000 = 0,05. Cioè, la probabilità di un crossing-over tra i geni A/a e B/b era 0,05, o il 5 percento. Un tale risultato indicherebbe che i geni erano definitivamente collegati, ma che erano abbastanza distanti tra loro perché occasionalmente si verificassero dei crossing-over. Sturtevant divise la sua mappa genetica in unità di mappa, o centimorgani (cM) , in cui una frequenza di ricombinazione di 0,01 corrisponde a 1 cM.
Rappresentando gli alleli in una mappa lineare, Sturtevant ha suggerito che i geni possono variare dal collegamento perfetto (frequenza di ricombinazione = 0) allo scollegamento perfetto (frequenza di ricombinazione = 0,5) quando i geni si trovano su cromosomi diversi o i geni si separano molto distanti sullo stesso cromosoma. I geni perfettamente scollegati corrispondono alle frequenze che Mendel ha previsto di assortire indipendentemente in un incrocio diibrido. Una frequenza di ricombinazione di 0,5 indica che il 50 percento della prole è ricombinante e l'altro 50 percento è di tipo parentale. Vale a dire, ogni tipo di combinazione di alleli è rappresentato con la stessa frequenza. Questa rappresentazione ha permesso a Sturtevant di calcolare in modo additivo le distanze tra diversi geni sullo stesso cromosoma. Tuttavia, man mano che le distanze genetiche si avvicinavano a 0,50, le sue previsioni diventavano meno accurate perché non era chiaro se i geni fossero molto distanti sullo stesso cromosoma o su cromosomi diversi.
Nel 1931, Barbara McClintock e Harriet Creighton dimostrarono il crossover di cromosomi omologhi nelle piante di mais. Settimane dopo, Curt Stern dimostrò la ricombinazione omologa microscopica in Drosophila . Stern osservò diversi fenotipi legati all'X che erano associati a una coppia di cromosomi X strutturalmente insolita e dissimile in cui a una X mancava un piccolo segmento terminale e l'altra X era fusa a un pezzo del cromosoma Y. Incrociando mosche, osservando la loro prole e quindi visualizzando i cromosomi della prole, Stern dimostrò che ogni volta che la combinazione di alleli della prole deviava da una delle combinazioni parentali, si verificava uno scambio corrispondente di un segmento del cromosoma X. L'uso di mosche mutanti con cromosomi X strutturalmente distinti era la chiave per osservare i prodotti della ricombinazione perché il sequenziamento del DNA e altri strumenti molecolari non erano ancora disponibili. Ora sappiamo che i cromosomi omologhi scambiano regolarmente segmenti nella meiosi, rompendo e riunendo reciprocamente il loro DNA in punti precisi. Aurora Ruiz-Herrera, ad esempio, studia il verificarsi di punti di rottura genetici in punti dei cromosomi noti come siti fragili. Identificando i siti fragili cromosomici condivisi tra umani e altri primati, Ruiz-Herrera ha fornito una comprensione più approfondita dell'evoluzione dei mammiferi e in particolare dell'evoluzione umana.
Collegamento all'apprendimento
[modifica | modifica sorgente]Rivedi qui il processo di Sturtevant per creare una mappa genetica sulla base delle frequenze di ricombinazione .
I caratteri mappati di Mendel si trovano su cromosomi diversi
[modifica | modifica sorgente]La ricombinazione omologa è un processo genetico comune, ma Mendel non l'ha mai osservato. Se avesse studiato sia i geni collegati che quelli non collegati, sarebbe stato molto più difficile per lui creare un modello unificato dei suoi dati sulla base di calcoli probabilistici. I ricercatori che da allora hanno mappato i sette tratti che Mendel ha studiato sui sette cromosomi del genoma di una pianta di pisello hanno confermato che tutti i geni da lui esaminati sono su cromosomi separati o sono sufficientemente distanti da essere statisticamente non collegati. Alcuni hanno suggerito che Mendel è stato enormemente fortunato a selezionare solo geni non collegati; mentre altri si chiedono se Mendel abbia scartato dati che suggerissero un collegamento. In ogni caso, Mendel ha costantemente osservato un assortimento indipendente perché ha esaminato geni che erano effettivamente non collegati.
Basi cromosomiche delle malattie ereditarie
[modifica | modifica sorgente]I disturbi ereditari possono insorgere quando i cromosomi si comportano in modo anomalo durante la meiosi. Possiamo dividere i disturbi cromosomici in due categorie: anomalie nel numero di cromosomi e riarrangiamenti strutturali cromosomici. Poiché anche piccoli segmenti cromosomici possono estendersi su molti geni, i disturbi cromosomici sono tipicamente drammatici e spesso fatali.
Identificazione dei cromosomi
[modifica | modifica sorgente]L'isolamento dei cromosomi e l'osservazione microscopica costituiscono la base della citogenetica e sono il metodo principale con cui i medici rilevano anomalie cromosomiche negli esseri umani. Un cariotipo è il numero e l'aspetto dei cromosomi e include la loro lunghezza, il modello di bande e la posizione del centromero. Per ottenere una visione del cariotipo di un individuo, i citologi fotografano i cromosomi e poi tagliano e incollano ogni cromosoma in un grafico, o cariogramma . Un altro nome è ideogramma ( Figura sotto).

In una data specie, possiamo identificare i cromosomi in base al loro numero, dimensione, posizione del centromero e schema di bande. In un cariotipo umano, gli autosomi o "cromosomi del corpo" (tutti i cromosomi non sessuali) sono generalmente organizzati in ordine approssimativo di dimensione dal più grande (cromosoma 1) al più piccolo (cromosoma 22). I cromosomi X e Y non sono autosomi. Tuttavia, il cromosoma 21 è in realtà più corto del cromosoma 22. I ricercatori lo hanno scoperto dopo aver chiamato la sindrome di Down trisomia 21, riflettendo come questo disturbo derivi dal possesso di un cromosoma 21 in più (tre in totale). Non volendo cambiare il nome di questo importante disturbo, gli scienziati hanno mantenuto la numerazione del cromosoma 21 nonostante lo descrivano come avente il set di cromosomi più corto. Possiamo designare le "braccia" dei cromosomi che si proiettano da entrambe le estremità del centromero come corte o lunghe, a seconda delle loro lunghezze relative. Abbreviamo il braccio corto p (per "petite"); mentre, abbreviamo il braccio lungo q (perché segue "p" in ordine alfabetico). I numeri suddividono ulteriormente e denotano ogni braccio. Utilizzando questo sistema di denominazione, possiamo descrivere le posizioni dei cromosomi in modo coerente nella letteratura scientifica.
I genetisti usano i cariotipi per identificare le alterazioni cromosomiche
[modifica | modifica sorgente]Sebbene Mendel venga definito il "padre della genetica moderna", egli ha eseguito i suoi esperimenti senza nessuno degli strumenti che i genetisti di oggi impiegano di routine. Una di queste potenti tecniche citologiche è il cariotipo, un metodo con cui i genetisti possono identificare tratti caratterizzati da anomalie cromosomiche da una singola cellula. Per osservare il cariotipo di un individuo, un genetista raccoglie prima le cellule di una persona (come i globuli bianchi) da un campione di sangue o da altri tessuti. In laboratorio, il genetista stimola le cellule isolate a iniziare a dividersi attivamente. Il genetista applica quindi la colchicina chimica alle cellule per arrestare i cromosomi condensati in metafase. Il genetista induce quindi il rigonfiamento nelle cellule utilizzando una soluzione ipotonica in modo che i cromosomi si dividano. Infine, il genetista conserva il campione in un fissativo e lo applica su un vetrino.
Il genetista colora quindi i cromosomi con uno dei vari coloranti per visualizzare meglio i distinti e riproducibili modelli di bande di ogni coppia di cromosomi. Dopo la colorazione, il genetista visualizza i cromosomi utilizzando la microscopia a campo chiaro. Una scelta comune di colorazione è la colorazione Giemsa. La colorazione Giemsa produce circa 400-800 bande (di DNA strettamente avvolto e proteine condensate) disposte lungo tutte le 23 coppie di cromosomi. Un genetista esperto può identificare ogni banda. Oltre ai modelli di bande, i genetisti identificano ulteriormente i cromosomi in base alle dimensioni e alla posizione del centromero. Per ottenere la classica rappresentazione del cariotipo in cui le coppie di cromosomi omologhi si allineano in ordine numerico dal più lungo al più corto, il genetista ottiene un'immagine digitale, identifica ogni cromosoma e dispone manualmente i cromosomi in questo modello ( vedi galleria sotto).
Nella sua forma più elementare, il cariotipo può rivelare anomalie genetiche in cui un individuo ha troppi o troppo pochi cromosomi per cellula. Esempi di ciò sono la sindrome di Down, che si identifica tramite una terza copia del cromosoma 21, e la sindrome di Turner, che è caratterizzata dalla presenza di un solo cromosoma X nelle femmine invece dei normali due. I genetisti possono anche identificare grandi delezioni o inserzioni del DNA. Ad esempio, i genetisti possono identificare la sindrome di Jacobsen, che comporta tratti facciali distintivi e difetti cardiaci ed emorragici, tramite una delezione sul cromosoma 11. Infine, il cariotipo può individuare le traslocazioni , che si verificano quando un segmento di materiale genetico si stacca da un cromosoma e si riattacca a un altro cromosoma o a una parte diversa dello stesso cromosoma. Le traslocazioni sono implicate in alcuni tumori, tra cui la leucemia mieloide cronica.
Durante la vita di Mendel, l'ereditarietà era un concetto astratto che si poteva dedurre solo eseguendo incroci e osservando i tratti espressi dalla prole. Osservando un cariotipo, i genetisti di oggi possono effettivamente visualizzare la composizione cromosomica di un individuo per confermare o prevedere anomalie genetiche nella prole, persino prima della nascita.
-
Cariotipo umano: che sesso è?
-
Cariotipo umano
-
cariotipo umano con sindrome
-
Cariotipo di un individuo con sindrome di Down
Alterazioni del numero dei cromosomi
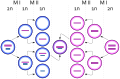
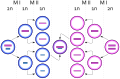
[modifica | modifica sorgente]Di tutti i disturbi cromosomici, le anomalie del numero dei cromosomi sono le più chiaramente identificabili da un cariotipo. I disturbi del numero dei cromosomi includono la duplicazione o la perdita di interi cromosomi, nonché cambiamenti nel numero di set completi di cromosomi. Sono causati dalla non disgiunzione , che si verifica quando coppie di cromosomi omologhi o cromatidi fratelli non riescono a separarsi durante la meiosi. Una sinapsi disallineata o incompleta, o una disfunzione dell'apparato del fuso che facilita la migrazione dei cromosomi, possono causare la non disgiunzione. Il rischio che si verifichi la non disgiunzione aumenta con l'età dei genitori.
La non disgiunzione può verificarsi durante la meiosi I o II, con risultati diversi ( Figura sotto). Se i cromosomi omologhi non riescono a separarsi durante la meiosi I, il risultato sono due gameti privi di quel particolare cromosoma e due gameti con due copie cromosomiche. Se i cromatidi fratelli non riescono a separarsi durante la meiosi II, il risultato è un gamete privo di quel cromosoma, due gameti normali con una copia cromosomica e un gamete con due copie cromosomiche.

| Esercizio |
|---|
Quale delle seguenti affermazioni sulla non disgiunzione è vera?
|
Aneuploidia
[modifica | modifica sorgente]Gli scienziati chiamano euploide un individuo con il numero appropriato di cromosomi per la sua specie . Negli esseri umani, l'euploidia corrisponde a 22 coppie di autosomi e una coppia di cromosomi sessuali. Un individuo con un errore nel numero di cromosomi è descritto come aneuploide , un termine che include monosomia (perdita di un cromosoma) o trisomia (acquisizione di un cromosoma estraneo). Gli zigoti umani monosomici a cui manca una qualsiasi copia di un autosoma non riescono invariabilmente a svilupparsi fino alla nascita perché mancano di geni essenziali. Ciò sottolinea l'importanza del "dosaggio genico" negli esseri umani. Anche la maggior parte delle trisomie autosomiche non riesce a svilupparsi fino alla nascita; tuttavia, le duplicazioni di alcuni cromosomi più piccoli (13, 15, 18, 21 o 22) possono dare origine a una prole che sopravvive per diverse settimane o molti anni. Gli individui trisomici soffrono di un diverso tipo di squilibrio genetico: un eccesso di dose genica. Gli individui con un cromosoma in più possono sintetizzare un'abbondanza di prodotti genici, che quel cromosoma codifica. Questa dose extra (150 percento) di geni specifici può portare a una serie di sfide funzionali e spesso impedisce lo sviluppo. La trisomia più comune tra le nascite vitali è quella del cromosoma 21, che corrisponde alla sindrome di Down. Bassa statura e dita stentate, distinzioni facciali che includono un cranio largo e una lingua grande e significativi ritardi dello sviluppo caratterizzano gli individui con questo disturbo ereditario. Possiamo correlare l'incidenza della sindrome di Down con l'età materna. Le persone anziane hanno maggiori probabilità di rimanere incinte con feti portatori del genotipo della trisomia 21 ( Figura in galleria).
-
La sindrome di Down
-
La trisomia 21 causa la sindrome di Down
-
L'incidenza di avere un feto affetto da trisomia 21 aumenta drasticamente con l'età materna
Poliploidia
[modifica | modifica sorgente]Chiamiamo poliploide un individuo con un numero di cromosomi superiore a quello corretto (due per le specie diploidi) . Ad esempio, fecondare un uovo diploide anomalo con uno spermatozoo aploide normale produrrebbe uno zigote triploide. Gli animali poliploidi sono estremamente rari, con solo pochi esempi tra i vermi piatti, i crostacei, gli anfibi, i pesci e le lucertole. Gli animali poliploidi sono sterili perché la meiosi non può procedere normalmente e invece produce principalmente cellule figlie aneuploidi che non possono produrre zigoti vitali. Raramente, gli animali poliploidi possono riprodursi asessualmente per aplodiploidia, in cui un uovo non fecondato si divide mitoticamente per produrre prole. Al contrario, la poliploidia è molto comune nel regno vegetale e le piante poliploidi tendono a essere più grandi e più robuste degli euploidi della loro specie ( Figura sotto).

Non disgiunzione dei cromosomi sessuali negli esseri umani
[modifica | modifica sorgente]Gli esseri umani mostrano effetti deleteri drammatici con trisomie e monosomie autosomiche. Pertanto, può sembrare controintuitivo che le femmine e i maschi umani possano funzionare normalmente, nonostante portino numeri diversi del cromosoma X. Piuttosto che un guadagno o una perdita di autosomi, si verificano variazioni nel numero di cromosomi sessuali con effetti relativamente lievi. In parte, ciò accade a causa del processo molecolare di inattivazione del cromosoma X. All'inizio dello sviluppo, quando gli embrioni di mammiferi femminili sono costituiti da poche migliaia di cellule (rispetto ai trilioni nel neonato), un cromosoma X in ogni cellula si inattiva condensandosi strettamente in una struttura quiescente (dormiente), o un corpo di Barr. La possibilità che un cromosoma X (derivato dalla madre o dal padre) si inattivi in ogni cellula è casuale, ma una volta che ciò si verifica, tutte le cellule derivate da quello avranno lo stesso cromosoma X inattivo o corpo di Barr. Con questo processo, le femmine compensano la loro doppia dose genetica di cromosoma X. Nei cosiddetti gatti "tartaruga", osserviamo l'inattivazione embrionale della X come variegatura del colore ( Figura sotto). Le femmine eterozigoti per un gene del colore del mantello legato alla X esprimeranno uno dei due diversi colori del mantello su diverse regioni del loro corpo, corrispondenti a qualsiasi cromosoma X inattivato nel progenitore cellulare embrionale di quella regione.

Un individuo portatore di un numero anomalo di cromosomi X inattiverà tutti i cromosomi X tranne uno in ciascuna delle sue cellule. Tuttavia, anche i cromosomi X inattivati continuano a esprimere alcuni geni e i cromosomi X devono riattivarsi per la corretta maturazione delle ovaie femminili. Di conseguenza, le anomalie dei cromosomi X si verificano in genere con lievi disturbi o disabilità intellettive e fisiche, nonché sterilità. Se il cromosoma X è completamente assente, l'individuo non si svilupperà nell'utero.
Gli scienziati hanno identificato e caratterizzato diversi errori nel numero dei cromosomi sessuali. Gli individui con tre cromosomi X, triplo-X, sono fenotipicamente femmine ma esprimono ritardi nello sviluppo e ridotta fertilità. Il genotipo XXY, corrispondente a un tipo di sindrome di Klinefelter, corrisponde a individui fenotipicamente maschi con testicoli piccoli, seni ingrossati e peli corporei ridotti. Esistono tipi più complessi di sindrome di Klinefelter in cui l'individuo ha fino a cinque cromosomi X. In tutti i tipi, ogni cromosoma X tranne uno subisce inattivazione per compensare l'eccesso di dosaggio genetico. Noi vediamo questo come diversi corpi di Barr in ogni nucleo cellulare. La sindrome di Turner, caratterizzata da un genotipo X0 (vale a dire, un solo cromosoma sessuale), corrisponde a un individuo fenotipicamente femmina con bassa statura, pelle palmata nella regione del collo, problemi di udito, cardiaci e sterilità.
-
Nascita di una cellula con cariotipo XXY dovuta a una non disgiunzione di un cromosoma X da un cromosoma Y durante la meiosi I del maschio.
-
Nascita di una cellula con cariotipo XXY dovuta a una non disgiunzione di un cromosoma X durante la meiosi II della femmina.
-
Cariotipo di un Klinefelter
-
La sindrome di Klinefelter
-
Monosomia X, sindrome di Turner
-
Cinque ragazze e donne ispaniche con sindrome di Turner , una condizione caratterizzata da una singola copia del cromosoma X
Alterazioni nella struttura del cromosoma
[modifica | modifica sorgente]Oltre a perdere o guadagnare un intero cromosoma, un segmento cromosomico può duplicarsi, perdersi o modificarsi.
Duplicazioni ed eliminazioni
[modifica | modifica sorgente]Le duplicazioni e le delezioni spesso producono prole che sopravvive ma presenta anomalie. I segmenti cromosomici duplicati possono fondersi con cromosomi esistenti o possono essere liberi nel nucleo. Cri-du-chat (dal francese per "grido del gatto") è una sindrome che si verifica con anomalie del sistema nervoso e caratteristiche fisiche identificabili che derivano da una delezione della maggior parte del 5p (il piccolo braccio del cromosoma 5) ( Figura sotto). I neonati con questo genotipo emettono un caratteristico pianto acuto su cui si basa il nome del disturbo.

Figura 13.10 Questa figura mostra un individuo con sindrome del cri-du-chat all'età di due, quattro, nove e 12 anni. (credito: Paola Cerruti Mainardi)
-
Mutazioni nel singolo cromosoma
Riarrangiamenti strutturali cromosomici
[modifica | modifica sorgente]I citologi hanno caratterizzato numerosi riarrangiamenti strutturali nei cromosomi, ma le inversioni e le traslocazioni cromosomiche sono le più comuni. Possiamo identificare entrambi durante la meiosi tramite l'appaiamento adattivo dei cromosomi riarrangiati con i loro precedenti omologhi per mantenere un allineamento genico appropriato. Se i geni su due omologhi non sono orientati correttamente, un evento di ricombinazione potrebbe causare la perdita di geni da un cromosoma e l'acquisizione di geni dall'altro. Ciò produrrebbe gameti aneuploidi.
Inversioni cromosomiche
[modifica | modifica sorgente]Un'inversione cromosomica è il distacco, la rotazione di 180° e il reinserimento di una parte di un cromosoma. Le inversioni possono verificarsi in natura come risultato di un taglio meccanico o dall'azione di elementi trasponibili (sequenze di DNA speciali in grado di facilitare la riorganizzazione di segmenti cromosomici con l'aiuto di enzimi che tagliano e incollano sequenze di DNA). A meno che non interrompano una sequenza genica, le inversioni cambiano solo l'orientamento genico e hanno probabilmente effetti più lievi degli errori aneuploidi. Tuttavia, l'orientamento genico alterato può causare cambiamenti funzionali perché i regolatori dell'espressione genica potrebbero spostarsi fuori posizione rispetto ai loro bersagli, causando livelli aberranti di prodotti genici.
-
schema delle mutazioni della struttura del cromosoma
L'inversione del cromosoma 18
[modifica | modifica sorgente]Non tutti i riarrangiamenti strutturali dei cromosomi producono individui non vitali, compromessi o sterili. In rari casi, un tale cambiamento può portare all'evoluzione di nuove specie. Infatti, un'inversione pericentrica nel cromosoma 18 sembra aver contribuito all'evoluzione umana. Questa inversione non è presente nei nostri parenti genetici più prossimi, gli scimpanzé. Umani e scimpanzé differiscono citogeneticamente per inversioni pericentriche su diversi cromosomi e per la fusione di due cromosomi separati negli scimpanzé che corrispondono al cromosoma due negli umani.
Gli scienziati ritengono che una inversione nel cromosoma 18 si sia verificata nei primi esseri umani in seguito alla loro divergenza da un antenato comune con gli scimpanzé circa cinque milioni di anni fa. I ricercatori che hanno caratterizzato questa inversione hanno suggerito che circa 19.000 basi nucleotidiche sono state duplicate su 18p e la regione duplicata è stata invertita e reinserita sul cromosoma 18 di un essere umano ancestrale.
Un confronto tra geni umani e scimpanzé nella regione di questa inversione indica che due geni, ROCK1 e USP14 , adiacenti sul cromosoma 17 dello scimpanzé (che corrisponde al cromosoma 18 umano) sono posizionati più distanti sul cromosoma 18 umano. Ciò suggerisce che uno dei punti di interruzione dell'inversione si è verificato tra questi due geni. È interessante notare che umani e scimpanzé esprimono USP14 a livelli distinti in specifici tipi di cellule, tra cui cellule corticali e fibroblasti. Forse l'inversione del cromosoma 18 in un umano ancestrale ha riposizionato geni specifici e ripristinato i loro livelli di espressione in modo utile. Poiché sia ROCK1 che USP14 codificano enzimi cellulari, un cambiamento nella loro espressione potrebbe alterare la funzione cellulare. Non sappiamo in che modo questa inversione abbia contribuito all'evoluzione degli ominidi, ma sembra essere un fattore significativo nella divergenza degli umani dagli altri primati. 1
Traslocazioni
[modifica | modifica sorgente]Una traslocazione si verifica quando un segmento cromosomico si dissocia e si riattacca a un cromosoma diverso, non omologo. Le traslocazioni possono essere benigne o avere effetti devastanti a seconda di come le posizioni dei geni vengono alterate rispetto alle sequenze regolatrici. In particolare, specifiche traslocazioni si sono verificate con diversi tumori e con la schizofrenia. Le traslocazioni reciproche derivano dallo scambio di segmenti cromosomici tra due cromosomi non omologhi in modo tale che non vi sia alcun guadagno o perdita di informazioni genetiche.

Genetica dei virus
[modifica | modifica sorgente]Vedi il paragrafo "Fasi delle infezioni da virus" e "Batteriofagi" nella pagina riguardante i virus
Genetica dei batteri
[modifica | modifica sorgente]Vedi nella pagina che parla dei batteri
Attività
[modifica | modifica sorgente]Esercizi di genetica su Wikiversità
Esercitazione di genetica su Wikiversità
Esercizi sugli alberi genealogici in inglese (genetica mendeliana e post-mendeliana) (su LibreTexts da KanAcademy)
Diversi tipi di esercizi con diverse difficoltà, ordinati per categoria, di genetica in generale, con soluzioni (su https://english.biologia-geologia.com/ con pubblicità)
Esercizi sulla genetica mendeliana in inglese (su OpenStax)
Esercizi un po' più difficili e di riflessione in inglese (su OpenStax)
Esercitazioni di genetica postmendeliana su Wikiversità
Esercizi sugli alberi genealogici in pdf in italiano (su Saccopedia)